Optimización de los protocolos de extracción de ADN y del marcador molecular tipo RAPD en Anonáceas
Optimization of the protocols of extraction of ADN and of the molecular marker type RAPD in Anonáceas
Yanet Alfonso Alonso,I M.Cs. Caridad Noriega Carrera,II Dra.C. Miriam Isidrón Pérez,I Lucy Andraca Collazo,I M.Cs. Dubiel Alfonso González,I Dra.C. Daymara Rodríguez Alfonso,I
IUniversidad
Agraria de La Habana, autopista nacional km 23 ½, carretera Tapaste, San José
de las Lajas, Mayabeque, Cuba. CP 32700.
IIEstación Experimental de Frutales de Alquízar, carretera de Pestana,
km 2 ½, Finca Reunión, Alquízar, Artemisa, Cuba.
RESUMEN
Las técnicas moleculares requieren de protocolos que permitan determinar los niveles de variación genética, dentro de las poblaciones en diferentes condiciones ambientales. Tanto la optimización del aislamiento del ADN, como el de las condiciones de trabajo de las amplificaciones, son fundamentales para alcanzar el éxito de los análisis moleculares, por lo que la presente investigación tiene como objetivo: optimizar los protocolos de extracción de ADN y del marcador molecular tipo RAPD (Random Amplified Polymorphic DNA) en Anonáceas. Para la extracción de ADN se utilizaron los Kit Nucleon PHYTOpure y DNeasy® de QIAGEN. Se ajustaron las condiciones de trabajo del protocolo de amplificación, en el que se variaron las concentraciones de ADN y las de los cebadores utilizados. Seguido de esto, se realizó un testaje con diez cebadores de la serie OPH y cinco de la serie OPA, para seleccionar los más polimórficos. Los primeros resultados con el Kit Nucleon PHYTO pure mostraron un ADN de baja calidad, debido a la alta fenolización del material vegetal, no así con el Kit DNeasy® de QIAGEN, que permitió obtener un ADN de calidad, pureza y homogeneidad a una concentración aproximada de 30 ng µL-1. Los mayores productos de amplificación se obtuvieron al usar 3 ng µL-1 de cebador y 2 ng µL-1 de ADN. Los cuatro cebadores que presentaron mayor polimorfismo fueron OPA-16, OPH-03, OPH-13 y OPH-18. Los resultados de esta investigación permitieron optimizar las condiciones de trabajo de la técnica RAPD para la caracterización de la colección ex situ de Anonáceas bajo nuestras condiciones ambientales.
Palabras clave: aislamiento de ADN, Annonaceae, RAPD.
ABSTRACT
The molecular techniques need of protocols that allow determine levels of genetic change inside the populations in different environmental conditions. So much the optimization of the isolation of the DNA, as that of the working conditions of the amplifications, they are fundamental to reach the success of the molecular analyses, therefore the present research has like objective: optimize protocols of extraction of DNA and of the molecular marker type RAPD (Random Amplified Polymorphic DNA) in Anonáceas. For the DNA extraction were used the Kit Nucleon PHYTOpure and DNeasy® of QIAGEN. The working conditions of the protocol of amplification were fittedand changed the concentrations of DNA and of the used chokers. Followed by this, a tes was realized with 10 fatteners of the series OPH and five of the series OPA, to select more polimorphiss. The first results obtained with the Kit Nucleon PHYTOpure showed an DNA of low quality, due to the high fenolization of the vegetable material, not like that with the Kit DNeasy® of QIAGEN, who allowed obtain an DNA of quality, purity and homogeneity to an approximate concentration of 30 ng µ L-1. The biggest amplification products were obtained touse 3 ng µ L-1 of choke and 2 ng µ L-1 of DNA. Four chokers that presented major polymorphism were OPA-16, OPH-03, OPH-13 and OPH-18. The results of this research allowed optimize the working conditions of the technical RAPD for the characterization of the collection ex-situ of Anonáceas under our environmental conditions.
Key words: DNA isolation, Annonaceae, RAPD.
INTRODUCCIÓN
La explotación
de las plantas ha impulsado a los agricultores a seleccionar y mejorar líneas,
cultivos o determinadas especies con características deseadas (1). En
la actualidad se ha recurrido a técnicas de biología molecular
con el fin de obtener marcadores genéticos específicos de cada
especie, encontrar diferencias polimórficas y proporcionar la información
necesaria para la identificación de materiales vegetales en estudioA
(2, 3, 4).
Los marcadores moleculares permiten estimar la distancia genética, la
identificación y la discriminación de poblaciones, variedades,
líneas puras e híbridos; además establecen relaciones de
parentesco y localizan e identifican regiones del ADN que afectan caracteres
cuantitativos (5, 6). El análisis de los marcadores genéticos
es muy útil por el polimorfismo que detectan, la herencia mendeliana
sin epístasis; es decir, sin interacción entre los genes, la insensibilidad
a los factores ambientales o al desarrollo de la planta y su fácil identificación
y co-dominancia (7, 8).
En la identificación de una especie, el primer paso a desarrollar es
el aislamiento del ADN o material genético, el cual debe estar lo suficientemente
puro para su manipulación y amplificación. Actualmente existen
diferentes sistemas comerciales que permiten la extracción del ADN a
partir del material vegetal. Sin embargo, no hay que descartar el uso de otras
técnicas que han sido propuestas con el fin de disminuir la interferencia
que provoca el alto contenido de polifenoles o polisacáridos en determinadas
especies como los Agaves y las Anonáceas. Esta interferencia en la muestra
trae consigo la disminución de la pureza y el rendimiento del ADNA,B.
Las técnicas moleculares requieren de protocolos que permitan determinar
los niveles de variación genética dentro de las poblaciones, en
diferentes condiciones ambientales y la caracterización de los recursos
fitogenéticos. Es esencial contar con métodos adecuados de aislamiento
y purificación del ADN que permita la aplicación de diversas técnicas
de biología molecular, así como de eficientes protocolos para
la amplificación del ADN mediante marcadores (9).
Existen dos importantes alternativas de conservación de los recursos
fitogenéticos (RFG) la in situ y la ex situ, en la
actualidad se destaca la última (10).
El éxito de este tipo de conservación depende de la accesibilidad
de los materiales conservados en ellos y la correcta caracterización
de su germoplasma (11). Esto facilita el manejo racional de las colecciones
y los programas de mejora, la selección adecuada de los progenitores
para estrategias de cruzamiento y la construcción de mapas genéticos.
Además, elimina las duplicaciones y asegura la correcta identificación
de sus accesiones (12, 13).
En Cuba, la única colección ex situ de Anonáceas
que existe se ecuentra ubicada en la Estación Experimental de Frutales
de Alquízar, perteneciente al Instituto de Investigaciones de Fruticultura
Tropical (IIFT). Esta familia comprende un importante grupo de frutas tropicales
muy apetecidas por la población. Sin embargo, las plantaciones comerciales
establecidas se han reducido notablemente, su cultivo se ha visto limitado por
años, por lo que solo se les localiza casi exclusivamente en los patios
y jardines de los poblados rurales y en algunas ciudades (14, 15).
En el país no existen informes sobre el uso de marcadores moleculares
tipo RAPD para la caracterización de la familia Annonaceae.
El éxito, tanto de esta técnica molecular como de otras, radica
en el ajuste de las condiciones de trabajo, desde el aislamiento de ADN hasta
las condiciones de la PCR, por esta razón, la presente investigación
tiene como objetivo optimizar los protocolos de extracción de ADN y del
marcador molecular tipo RAPD en Anonáceas.
MATERIALES Y MÉTODOS
El presente trabajo
se realizó a partir de muestras de plantas de la colección ex
situ de la familia Annonaceae de Cuba. La colección se
encuentra conservada en la Estación Experimental de Frutales de Alquízar,
perteneciente al Instituto de Investigaciones de Fruticultura Tropical (IIFT),
ubicada en los 22° 47 de latitud Norte y los 82° 31 de longitud Oeste
a 110 m s.n.m. Se encuentra sembrada en un suelo Ferralítico Rojo típico
con pH alrededor de 5,5-6,5 y topografía llana, a una distancia de plantación
de 7 x 7 m. No presenta riego, por lo que está bajo condiciones de secano,
sin fertilización y los árboles tienen diferentes edades (plantas
de resiembra y hasta con más de 15 años).
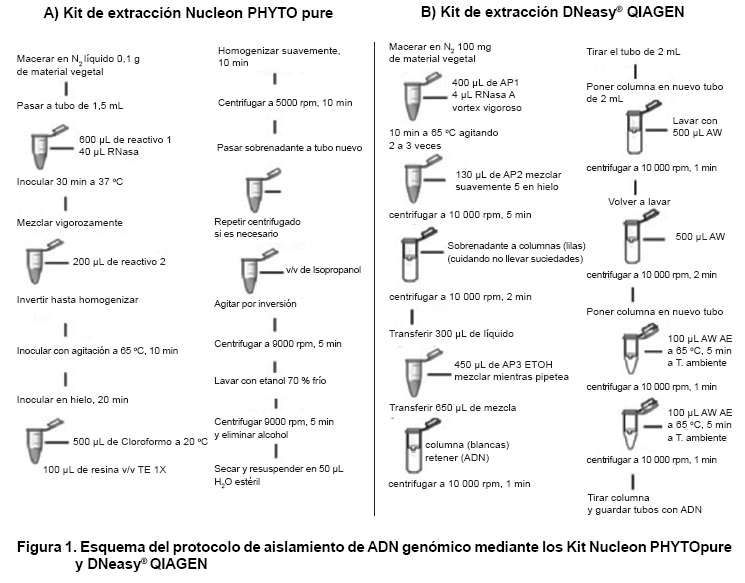
Aislamiento de ADN
Para la optimización del protocolo de amplificación del ADN con
el marcador molecular tipo, se garantizó la calidad y la homogeneidad
del ADN de los materiales. En el aislamiento del ADN se emplearon dos protocolos,
el Kit de extracción NucleonPHYTOpure (sin columnas) de la firma comercial
Amersham y el Kit de extracción DNeasy® de la firma comercial
QIAGEN (con columnas) Figura
1. Como material vegetal se utilizaron hojas jóvenes de plantas visiblemente
sanas (Tabla I).

La calidad y homogeneidad del ADN genómico se verificó en gel
de agarosa al 0,8 % en buffer TBE al 1X, que se corrió a 100 V durante
30 min. Para estimar visualmente la concentración del ADN el gel se observó
en un transiluminador de luz ultravioleta.
Amplificación del ADN con el marcador molecular tipo RAPD
Para establecer las condiciones de trabajo en la optimización del protocolo
a utilizar en la amplificación del ADN mediante el marcador molecular
tipo RAPD, se modificaron algunos de los componentes de la mezcla maestra para
un volumen total de 30 µL. Inicialmente se probaron diferentes concentraciones
de ADN genómico (30 ng µL-1) (1, 2 y 4 ng µL-1
de reacción), una vez ajustada la concentración más adecuada,
se procedió entonces a variar la del cebador (100 µM) (1, 2 y 3
ng µL-1) (Tabla II).
En
la amplificación por PCR se utilizó un termociclador de la marca
TECHNE modelo TC-3000 en el que se siguió un programa de tres horas de
duración aproximadamente donde la desnaturalización inicial fue
de 5 min a 94 °C; seguida de 35 ciclos compuestos cada ciclo de desnaturalización
de 30 s a 94 °C, hibridación a 36 °C durante 30 s y extensión
a 72 °C durante 1 min, el programa finaliza con un ciclo de extensión
final a 72 °C durante 5 min.
Los productos de amplificación RAPD generados se separaron mediante electroforesis
horizontal, en geles de agarosa al 1,5 % (buffer 1X TBE). Se corrieron en cámara
electroforética con buffer de conducción eléctrica a igual
concentración, a 80 V y se utilizó el marcador de pesos moleculares
(1Kb DNA Ladder, INVITROGEN®).
Estos geles, se tiñeron con Bromuro de etidio 0,75 µg mL-1
y se observaron en un transiluminador de luz ultravioleta (312 nm), para la
visualización de los fragmentos amplificados.
Una vez estandarizado el protocolo de amplificación del ADN, mediante
el marcador molecular tipo RAPD, se procedió a la selección de
los cebadores más polimórficos para la posterior caracterización
de la colección ex situ de Anonáceas. Para esto se probaron
un total de 15 cebadores decaméricos (Tabla III) de
Operon Technologies (Alameda CA, EE.UU.) con el ADN de tres genotipos diferentes
de las accesiones en estudio.
La mezcla maestra que se utilizó fue la que permitió mejor amplificación del ADN, donde en un volumen total de 30 µL habían 15 µL de Master mix (2X), 3 µL de cebador (10 pmoles µL-1); 0,2 µL de Taq polimerasa (5 U µL-1) (Fermentas), 30 ng de ADN genómico y a completar con agua. En la amplificación de la PCR para la separación de los productos RAPD y su visualización, se siguió la metodología anteriormente descrita. De los productos amplificados, se tomaron fotografías de 300 píxeles/pulgadas y se analizaron manualmente para seleccionar los cebadores que mayor polimorfismo detectaron (las reacciones de PCR se repitieron dos veces).
RESULTADOS Y DISCUSIÓN
Aislamiento de ADN
En la selección del protocolo adecuado de aislamiento de ADN, no se obtuvieron
los resultados deseados con el Kit de extracción Nucleon PHYTOpure (Figura
2). Este resultado negativo pude deberse a los elevados niveles de fenolización
que se alcanzaron en la mayoría de las accesiones, fundamentalmente en
las muestras de la especie A. reticulata. Resultados similares de fenolización
han sido obtenidos al extraer el ADN de plantas de Anonáceas conservadas
en el banco de germoplasma de EcuadorC
y en Annona senegalensis Pers recolectadas en un bosque de Kachia Kaduna,
Nigeria (15).

Esta es una etapa fundamental en el proceso de amplificación del ADN
mediante PCR, ya que la presencia de polifenoles, polisacáridos, proteínas,
taninos, pigmentos, entre otros; son elementos que dañan el ADN, su precipitación
y como consecuencia de ello comprometen posteriores análisis moleculares
(9, 16).
La ausencia del ADN también podría deberse a que en este Kit no
se utilizan columnas, por lo que el tiempo de exposición del material
vegetal y su manipulación fue mayor. Aunque los Kit facilitan el aislamiento
del ADN genómico y disminuyen el tiempo de manipulación se ha
demostrado, en Tectona grandis L., que la carencia de columnas en estos permite
obtener mejores resultados (17), respecto a los métodos clásicos
con CTAB (18, 19, 20), empleados en plantas, aunque no superan la calidad que
se obtiene con aquellos que si tienen incorporadas columnas.
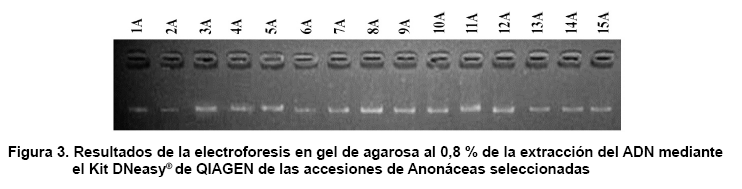
Por otra parte, se realizó un nuevo aislamiento de ADN mediante Kit de
extracción DNeasy® de QIAGEN, con el que se alcanzaron excelentes
resultados (Figura 3),
pues fue posible obtener un ADN genómico de calidad, pureza y homogeneidad.
Esto en gran medida puede deberse a las facilidades y a la calidad que permiten
el uso de las columnas. A pesar de los altos costos de estos Kit de extracción
de ADN, las grandes ventajas, en cuanto a la obtención de un ADN de calidad,
la disminución de la manipulación y el menor número de
equipos utilizados, han sido demostrados en especímenes de la familia
Juncaceae (21).
Las columnas del kit DNeasy® utilizado en este estudio, tienen como
características, según QIAGEND,
el intercambio aniónico. Se plantea que esta propiedad le permite tener
una alta afinidad por ácidos nucleicos, junto con el uso de sales caotrópicas
que rompen puentes de hidrógeno, aumentando la solubilidad de sustancias
no polares en agua, las que afectan fundamentalmente la estructura secundaria
de polímeros tales como ADN, ARN y proteínas (17).
La calidad del ADN y la homogeneidad entre las muestras son condiciones fundamentales
para la amplificación del mismo (22). Por otra parte, se plantea que
la calidad y la pureza de los ácidos nucleicos son dos de los elementos
más importantes en los análisis moleculares y la clonaciónE.
Otro criterio, es que contar con protocolos rápidos, fiables y de bajo
costo para la extracción de ADN resulta siempre deseable (9).
Amplificación del ADN con el marcador molecular tipo RAPD
De las tres concentraciones de ADN genómico utilizadas (1, 2, 4 ng µL-1)
con los tres genotipos seleccionados para establecer las modificaciones más
adecuadas, los mejores resultados se fueron al usar 2 ng µL-1
y aceptables con 1 ng µL-1. Con esta primera se alcanzó
un mayor número de bandas (Figura
4).
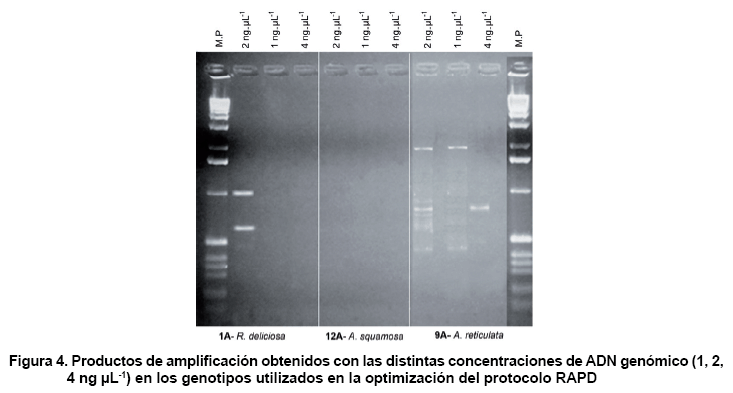
Como se observa en la figura
anterior, con las tres concentraciones de ADN genómico empleadas no se
generaron productos de amplificación (bandas) en las amplificaciones
de A. squamosa, por tal motivo, se decidió eliminarlas de los
restantes análisis. Una de las causas de la ausencia de bandas son que
los cebadores utilizados no encontraron zonas de homología del ADN o
la calidad del ADN genómico en esta especie.
Aunque en los trabajos de optimización de protocolos con el marcador
molecular tipo RAPD, este no es uno de los componentes que se tiende a variar,
es necesario ajustar la concentración a utilizar porque afecta las amplificaciones.
Los rangos de concentración de ADN más recomendados a usar deben
estar entre los 50-100 ng, ya que por encima de este valor se afecta la amplificación
por la dilución de los cebadores y por debajo no se obtiene suficiente
producto de amplificación (23).
Otro de los componentes que se varían en la mezcla de amplificación
es la concentración del cebador. Entre las tres concentraciones utilizadas
en la reacción (1, 2 y 3 ng µL-1de reacción)
la mejor fue la última (Figura
5). Al aplicar 1 ng µL-1 de cebador no se obtuvo prácticamente
producto de la amplificación, solamente una banda en A. reticulata;
aunque con 2 ng µL-1 hay un incremento de productos de amplificación
este es inferior a lo obtenido con 3 ng µL-1, en el que el
patrón de bandas es más característico de un marcador RAPD.
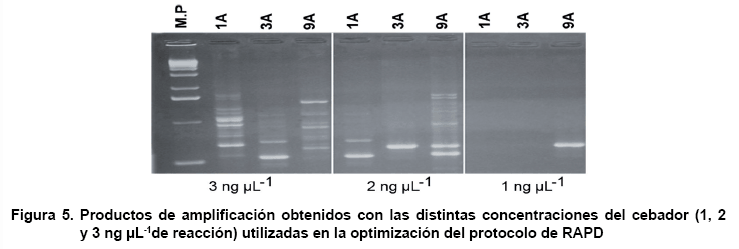
No hay una concentración de cebadores establecida para las amplificaciones,
este es un elemento que se ajusta en cada especie y en condiciones diferentes
de trabajo (9). Estos mismos autores, pero en la especie Ceratozamia mexicana
Brongn. emplearon dosis de cebadores de 1, 2 y 3 ng µL-1; sin
embargo, con el primero es suficiente para obtener bandas claras, distintivas
y reproducibles.
El ADN de las tres accesiones seleccionadas para el pesquizaje amplificó
con los 15 cebadores probados. Los productos de amplificación de estas
tres especies, revelaron en los geles para cada una, entre dos y cinco bandas
polimórficas (datos no mostrados), lo que se corroboró en ambas
réplicas. Finalmente fueron seleccionados los cuatro cebadores más
polimórficos: OPA-16, OPH-03, OPH-13 y OPH-18, a partir de los cuales
podrá ser estudiada la diversidad genética de la colección.
CONCLUSIONES
Nota al Pie
AFeria,
R.I.A. Clonación y caracterización de fragmentos RAPD y loci
microsatélite, asociados a Psidium guajava L. cultivada en 4 estados
de la República Mexicana. [en línea] [Tesis de Doctorado],
Universidad Autónoma Metropolitana, México, 2008, [Consultado:
28 marzo 2015], Disponible en: http://148.206.53.84/tesiuami/UAMI14289.pdf.
BHerrera,
R.J.L. Optimización de protocolos para la extracción de ADN
y uso del marcador SCAR ISPJ1 en Piñón (Jatropha curcas L.)
[en línea] [Tesis presentada en opción al título de Ingeniero
Agropecuario], Escuela Agrícola Panamericana El Zamorano, Honduras, 2010,
66 p., [Consultado: 28 marzo 2015], Disponible en: http://bdigital.zamorano.edu/handle/11036/601.
CEscribano,
M. Caracterización de marcadores moleculares para la identificación
de genotipos, estudio de diversidad genética y mejora del chirimoyo (A.
cherimola Mill.). [Tesis de Doctorado], Facultad de Ciencias, departamento
de microbiología. Universidad de Málaga, España, 2006,
139 p.
DQIAGEN.
QIAGEN® Product Guide Supplement [en línea],
2011, [Consultado: 28 marzo 2015], Disponible en: http://www.google.com.cu/url?sa=t&rct=j&q=&esrc=s&source=web&cd=1&ved=0CB4QFjAA&url=http%3A%2F%2Fwww.qiagen.com%2Fresources%2Fdownload.aspx%3Fid%3Dc6c86754-4ffb-49ef-b927-a5773d28d6e9%26lang%3Den&ei=UucWVenaHI_2yQTlj4CQDw&usg=AFQjCNFMYy7QNGOJPBtLctm5Lot5ddHLBg&bvm=bv.89381419,d.aWw&cad=rja.
ESomma,
M. Extracción y Purificación de ADN [en línea],
edit. Organización mundial de la salud Oficina Regional para Europa,
2011,Disponible en: http://www.google.com.cu/url?sa=t&rct=j&q=&esrc=s&source=web&cd=1&ved=0CB4QFjAA&url=http%3A%2F%2Fgmo-crl.jrc.ec.europa.eu%2Fcapacitybuilding%2Fmanuals%2FManual%2520ES%2FSesi%25C3%25B3n4.pdf&ei=-OgWVcvoHYazyASkvoKYBA&usg=AFQjCNGR15n6i7WIgQ41a05Spguu64H4GA&bvm=bv.89381419,d.aWw&cad=rja.
BIBLIOGRAFÍA
1. González,
L. M. ‘‘Aspectos generales sobre la tolerancia a la salinidad en las plantas
cultivadas’’. Cultivos Tropicales, vol. 23, no. 2, 2013, pp. 27–37,
ISSN 0258-5936.
2. Espósito, M. A.; Martin, E.; Cravero, V. P. y Cointry, E. L. ‘‘Uso
de marcadores morfológicos, bioquímicos y moleculares SRAP para
diferenciar variedades de Cynara cardunculus L. (Asteraceae)’’.
Revista de la Facultad de Ciencias Agrarias. Universidad Nacional de Cuyo,
vol. 43, no. 2, diciembre de 2011, pp. 35-45, ISSN 1853-8665.
3. Sánchez-Betancourt, E. y Zarantes, V. M. N. ‘‘Evaluación de
marcadores moleculares tipo SCAR para determinar sexo en plantas de papaya (Carica
papaya L.)’’. Revista Corpoica Ciencia y Tecnología Agropecuaria,
vol. 9, no. 2, 23 de diciembre de 2013, pp. 31-36,
ISSN 0122-8706.
4. van Zonneveld, M.; Dawson, I.; Thomas, E.; Scheldeman, X.; van Etten, J.;
Loo, J. y Hormaza, J. I. ‘‘Application of Molecular Markers in Spatial Analysis
to Optimize In Situ Conservation of Plant Genetic Resources’’ [en línea].
En: eds. Tuberosa R., Graner A., y Frison E., Genomics of Plant Genetic
Resources, edit. Springer Netherlands, Dordrecht, 2014, pp. 67-91, ISBN
978-94-007-7571-8, [Consultado: 8 de octubre de 2015], Disponible en:
<http://link.springer.com/10.1007/978-94-007-7572-5_4>.
5. Perera, M. F.; García, M. G.; Noguera, A. S.; Sepúlveda Tusek,
M.; Filippone, M. P. y Castagnaro, A. P. ‘‘Evaluación de la variación
somaclonal en vitroplantas de caña de azúcar mediante marcadores
moleculares’’. Revista industrial y agrícola de Tucumán,
vol. 87, no. 2, diciembre de 2010, pp. 13-21, ISSN 1851-3018.
6. Valdés de la Cruz, M.; González, C.; Lara, R. M.; Román,
M. I.; Hernández, Y.; Hernández, R. M.; Cabrera, M. y Torrecilla,
G. ‘‘Diversidad genética de especies silvestres del género Nicotiana
I: caracterización mediante marcadores bioquímicos’’. Revista
de Protección Vegetal, vol. 25, no. 2, agosto de 2010, pp. 88-97,
ISSN 1010-2752.
7. Bautista-Puga, M.; Vazquez-Garcia, L. M.; Leszczynska-Borys, H.; Borys, M.
W. y Arzate-Fernandez, A. M. ‘‘Characterization of aztec lily through morphological
and molecular markers’’. Agrociencia, vol. 45, no. 4, 2011, pp. 413–422,
ISSN 1405-3195.
8. Rojas, K. M.; Roa, M.; Briceño, I.; Guaneme, C. y Gómez, A.
‘‘Polimorfismos de 17 marcadores STR del cromosoma-Y en una muestra poblacional
del altiplano cundiboyacense’’. Colombia Médica, vol. 42, no.
1, marzo de 2011, pp. 88-97, ISSN 1657-9534.
9. Sanchez-Coello, N.; Luna-Rodriguez, M.; Vazquez-Torres, M.; Rafael Sanchez-Velasquez,
L.; Santana-Buzzy, N.; Octavio-Aguilar, P. y Georgina Iglesias-Andreu, L. ‘‘Optimization
of a protocol for dna isolation and issr-pcr amplification system for ceratozamia
mexicana brongn (Zamiaceae)’’. Revista Chapingo Serie Ciencias
Forestales y del Ambiente, vol. 18, no. 1, 2012, pp. 127–137, ISSN 2007-3828.
10. FAO. Normas para bancos de germoplasma de recursos fitogenéticos
para la alimentación y la agricultura. Edición revisada ed.,
edit. Organización de las Naciones Unidas para la Alimentación
y la Agricult, Roma, 2014, 126 p., ISBN 978-92-5-308262-9.
11. Lobo, M. A. y Medina, C. I. C. ‘‘Conservación de recursos genéticos
de la agrobiodiversidad como apoyo al desarrollo de sistemas de producción
sostenibles’’. Revista Corpoica Ciencia y Tecnología Agropecuaria,
vol. 10, no. 1, 24 de diciembre de 2013, pp. 33-42, ISSN 0122-8706.
12. Rao, V. R.; Brown, A. H. D. y Jackson, M. Managing Plant Genetic Diversity.
edit. CABI, 13 de diciembre de 2001, 510 p., ISBN 978-0-85199-886-2.
13. Cubero Salmeron, J. I. Introducción a la Mejora Genética
Vegetal [en línea]. 2.a ed., edit. Mundi-Prensa, Madrid,
España, 2003, ISBN 84-7114-813-7, [Consultado: 28 de marzo de 2015],
Disponible en: <http://www.sidalc.net/cgi-bin/wxis.exe/?IsisScript=LIBROS.xis&method
=post&formato=2&cantidad=1&expresion=mfn= 007740>.
14. Cota, L.; Vieira, F.; Melo J; Brandão, M.; Santana, K.; Guedes, M.
y Oliveira, D. ‘‘Genetic diversity of Annona crassiflora (Annonaceae)
in northern Minas Gerais State’’. Genetics and Molecular Research,
vol. 10, no. 3, 2011, pp. 2172-2180, ISSN 1676-5680, DOI 10.4238/vol10-3gmr1188.
15. Ukwubile, C. A. ‘‘Genomic DNA extraction method from Annona senegalensis
Pers. (Annonaceae) fruits’’. African Journal of Biotechnology,
vol. 13, no. 6, 5 de febrero de 2014, pp. 749-753, ISSN 1684-5315, DOI 10.5897/AJB12.2956.
16. Puchooa, D. ‘‘A simple, rapid and efficient method for the extraction of
genomic DNA from lychee (Litchi chinensis Sonn.)’’. African Journal
of Biotechnology, vol. 3, no. 4, 25 de octubre de 2004, pp. 253-255, ISSN
1684-5315, DOI 10.4314/ajb.v3i4.14954.
17. Quiala, E.; Valledor, L.; Hazbun, R.; Barbón, R.; Feria, M. de; Chávez,
M. y Rodríguez, R. ‘‘Estandarización de un protocolo de obtención
de ADN genómico para la cuantificación de 5mC en brotes epicórmicos
de Tectona grandis L’’. Biotecnología vegetal, vol.
8, no. 1, 2008, pp. 51–56, ISSN 2074-8647.
18. Dellaporta, S. L.; Wood, J. y Hicks, J. B. ‘‘A plant DNA minipreparation:
Version II’’. Plant Molecular Biology Reporter, vol. 1, no. 4, 1 de
septiembre de 1983, pp. 19-21, ISSN 0735-9640, 1572-9818, DOI 10.1007/BF02712670.
19. Doyle, J. J. ‘‘Isolation of plant DNA from fresh tissue’’. Focus,
vol. 12, 1990, pp. 13-15.
20. Aras, S.; Duran, A. y Yenilmez, G. ‘‘Isolation of DNA for RAPD analysis
from dry leaf material of some Hesperis L. specimens’’. Plant Molecular
Biology Reporter, vol. 21, no. 4, 3 de septiembre de 2012, pp. 461-462,
ISSN 0735-9640, 1572-9818, DOI 10.1007/BF02772597.
21. Drábková, L.; Kirschner, J. y Vlcek, C. ‘‘Comparison of seven
DNA extraction and amplification protocols in historical herbarium specimens
of juncaceae’’. Plant Molecular Biology Reporter, vol. 20,
no. 2, 3 de septiembre de 2012, pp. 161-175, ISSN 0735-9640, 1572-9818, DOI
10.1007/BF02799431.
22. Oropeza, M.; Marilyn R, E. A. y Vargas C, T. E. ‘‘Establecimiento de un
protocolo rapds eficiente para plantas de ñame’’. Agronomía
Tropical, vol. 56, no. 4, diciembre de 2006, pp. 601-606, ISSN 0002-192X.
23. Almeida, I. P.; Graterol, L. R. A.; Osorio, G.; Ramis, C.; Bedoya, Á.
M.; Figueroa, R.; Molina, S. y Infante, D. ‘‘Método modificado de obtención
de ADN genómico en orquídeas (Cattleya spp.) para amplificación
con marcadores moleculares’’. Bioagro, vol. 23, no. 1, 2011, pp. 27–34.
Recibido: 15 de
mayo de 2015
Aceptado: 18 de enero de 2016
Yanet Alfonso Alonso, Universidad Agraria de La Habana, autopista nacional km 231/2, carretera Tapaste, San José de las Lajas, Mayabeque, Cuba. CP 18 y 19. Email: yanet_alfonso@unah.edu.cu