http://dx.doi.org/ 10.13140/RG.2.1.1666.8406
Caracterización fenotípica y genética de cuatro especies silvestres del género Solanum, sección Lycopersicon
Phenotypic and genetic characterization of four species of the Solanum genera, Lycopersicon section
M.Cs. María N. Morales-Palacio,I Dr.C. Ángel R. Morales-Astudillo,I Dra.C. Adriana Artiles-Valor,II M.Cs. Yoamel Milián-García,III Dra.C. Georgina Espinosa-LópezIII
IIDENTIGEN. Laboratorio Biomolecular, Consorcio Torre 1, Azuay y Sucre, Loja, Ecuador.
IICentro de Investigaciones Pesqueras, 5ta y 246. Barlovento, Playa. CP 19100, Ciudad de la Habana, Cuba. Afiliación actual Miami Dade College, USA.
IIIFacultad de Biología, Universidad de la Habana, Ciudad de la Habana, Cuba.
RESUMEN
El tomate (Solanum lycopersicum L.) es la hortaliza de mayor importancia económica en todo el planeta y uno de los vegetales más consumidos en el mundo. Las especies silvestres emparentadas son nativas de diversos hábitats que van desde Ecuador, Perú, hasta Chile y han sido empleadas para generar variedades adaptadas a factores bióticos y abióticos específicos en todo el mundo. Con el objetivo de evaluar la variación morfológica y genética en la colección de germoplasma de la Universidad Nacional de Loja (UNL) en Ecuador, se seleccionaron cuatro especies silvestres: Solanum pimpinellifolium, Solanum neorickii, Solanum habrochaites, Solanum lycopersicum var. cerasiforme. Con los caracteres morfológicos (20 cuantitativos, 20 cualitativos) se detectaron diferencias fenotípicas en los componentes vegetativos y los relacionados con la flor y el fruto. Sólo S. habrochaites se diferenció sobre la base de estos caracteres. La diversidad y estructura genética de las especies se determinó con 17 loci microsatélites. A pesar de que ninguno de los índices de variabilidad mostró diferencias estadísticamente significativas, debido a la gran varianza que presentan, la especie S. neorickii mostró los menores valores de variabilidad genética. Las distancias individuales, el número de grupos estructurados genéticamente y la diferenciación genética (FST) fueron congruentes y revelaron cuatro grupos diferenciados correspondientes a cada especie estudiada.
Palabras clave: banco de germoplasma, Ecuador, microsatélites, tomate, variación genética.
ABSTRACT
The tomato (Solanum lycopersicum L.) is the most economically important vegetable worldwide and one of the most consumed vegetable in the world. It’s wild relatives are native of various habitats ranging from Ecuador, Peru, and Chile and have been employed to generate varieties adapted to specific biotic and abiotic factors worldwide. In order to evaluate the morphological and genetic variation in the germplasm collection at the National University of Loja (UNL) in Ecuador, four wild species were selected: Solanum pimpinellifolium, Solanum neorickii, Solanum habrochaites, Solanum lycopersicum var. cerasiforme. With the morphological variables (20 quantitative, 20 qualitative) phenotypic differences in vegetative components and related to flower and fruit were detected. Only S. habrochaites was differentiated based on these variables. The diversity and genetic structure of the species were evaluated with 17 microsatellite loci. In spite of none of the variability indexes showed statistically significant differences due to the large variance presented, the species S. neorickii exhibited the lowest genetic variability values. The individual genetic distances, the number of groups genetically structured and the genetic differentiation (FST) were congruent and revealed four groups corresponding to each species tested.
Key words: germplasm bank, Ecuador, microsatellites, tomato, genetic variation.
INTRODUCCIÓN
El tomate (Solanum lycopersicum L.) es considerado una de las hortalizas de mayor importancia en muchos países del mundo y constituye aproximadamente el 30 % del consumo de verduras de los países en desarrollo, lo que representa cerca del 65 % de la producción mundial (1).
El centro primario de origen del tomate y de mayor distribución de las especies silvestres emparentadas, comprende las regiones situadas a lo largo de la Cordillera de los Andes, en el territorio que hoy comparten Colombia, Ecuador, Perú, Bolivia y Chile, incluidas las Islas Galápagos (2), donde crecen espontáneamente las especies silvestres del género Solanum. Estas plantas viven en una gran variedad de hábitats, desde el nivel del mar en la costa árida del Pacífico, hasta sobre los 3300 m s. n. m. en numerosos valles del lado oeste de los Andes (3).
La forma más primitiva del tomate cultivado es la variedad botánica Solanum lycopersicum var. cerasiforme (tomate cereza), que habita las regiones tropicales y subtropicales de Ecuador y Perú (4) y desde donde fue probablemente difundida en épocas precolombinas a toda América tropical. En Ecuador se encuentran también en forma silvestre las especies S. neorickii y S. habrochaites, mientras que en Perú se hallan además S. chilense, S. chmielewskii, S. pennellii, S. peruvianum y en el norte de Chile, S. chilense, todas ellas con frutos de color verde. Las formas más evolucionadas, comestibles, de fruto rojo o amarillo en su madurez que se encuentran en forma silvestre en el Ecuador son S. cheesmaniae y S. pimpinellifolium, mientras que en el Perú está la última citada .
Las especies silvestres relacionadas con el tomate poseen atributos individuales de importancia potencial para el mejoramiento de las variedades cultivadas (5); sin embargo, antes de promover la utilización de las especies silvestres, es necesario conocer la variabilidad genética conservada en su hábitat natural, realizar su inventario, descripción y caracterización.
Gran número de accesiones de especies silvestres y variedades cultivadas de tomate se mantienen en bancos de germoplasma de todo el mundo. Los caracteres morfológicos permiten diferenciar accesiones, determinar materiales promisorios y su utilidad para el mejoramiento genético. Por otra parte, los marcadores moleculares como los microsatélites (SSR) son ventajosos en la diferenciación de accesiones, identificación de la variedad y trazabilidad del producto, ya que han demostrado ser altamente potentes para distinguir cultivares de tomate estrechamente relacionados (6–8), sin estar afectados por las condiciones ambientales. Las técnicas de caracterización morfológica y molecular se complementan entre sí, en la medida en que la diversidad molecular no considera las interacciones genotipo-ambiente, por ello ambos métodos en conjunto pueden proveer un conocimiento amplio y real de la diversidad de especies.
El banco de germoplasma del Centro de Biotecnología de la Universidad Nacional de Loja-Ecuador (UNL), posee alrededor de 2 000 accesiones del género Solanum. Este material ha sido caracterizado parcialmente a nivel morfológico, además de que en trabajos previos las accesiones ecuatorianas han sido tradicionalmente subrepresentadas. Este estudio propone caracterizar fenotípica y genéticamente cuatro especies silvestres del género Solanum, en su mayoría recolectadas en territorio ecuatoriano, con el objetivo de determinar la extensión de su variabilidad y establecer la utilidad de los marcadores moleculares en la diferenciación de las especies propuestas.
MATERIALES Y MÉTODOS
El material empleado en el presente estudio pertenece a la colección del banco de germoplasma del Centro de Biotecnología de la UNL. Procede de recolectas realizadas en una amplia área geográfica que comprende el centro primario de diversidad del tomate (Ecuador y Perú, Figura 1), ejecutadas por el equipo del Centro de Biotecnología de la UNL, en colaboración con el Parque Nacional Galápagos, el Centro de Conservación y Mejora de la Agrodiversidad Valenciana (COMAV) de la Universidad Politécnica de Valencia, la Universidad Nacional de Piura-Perú y la Universidad Nacional Pedro Ruíz Gallo de Lambayeque-Perú.
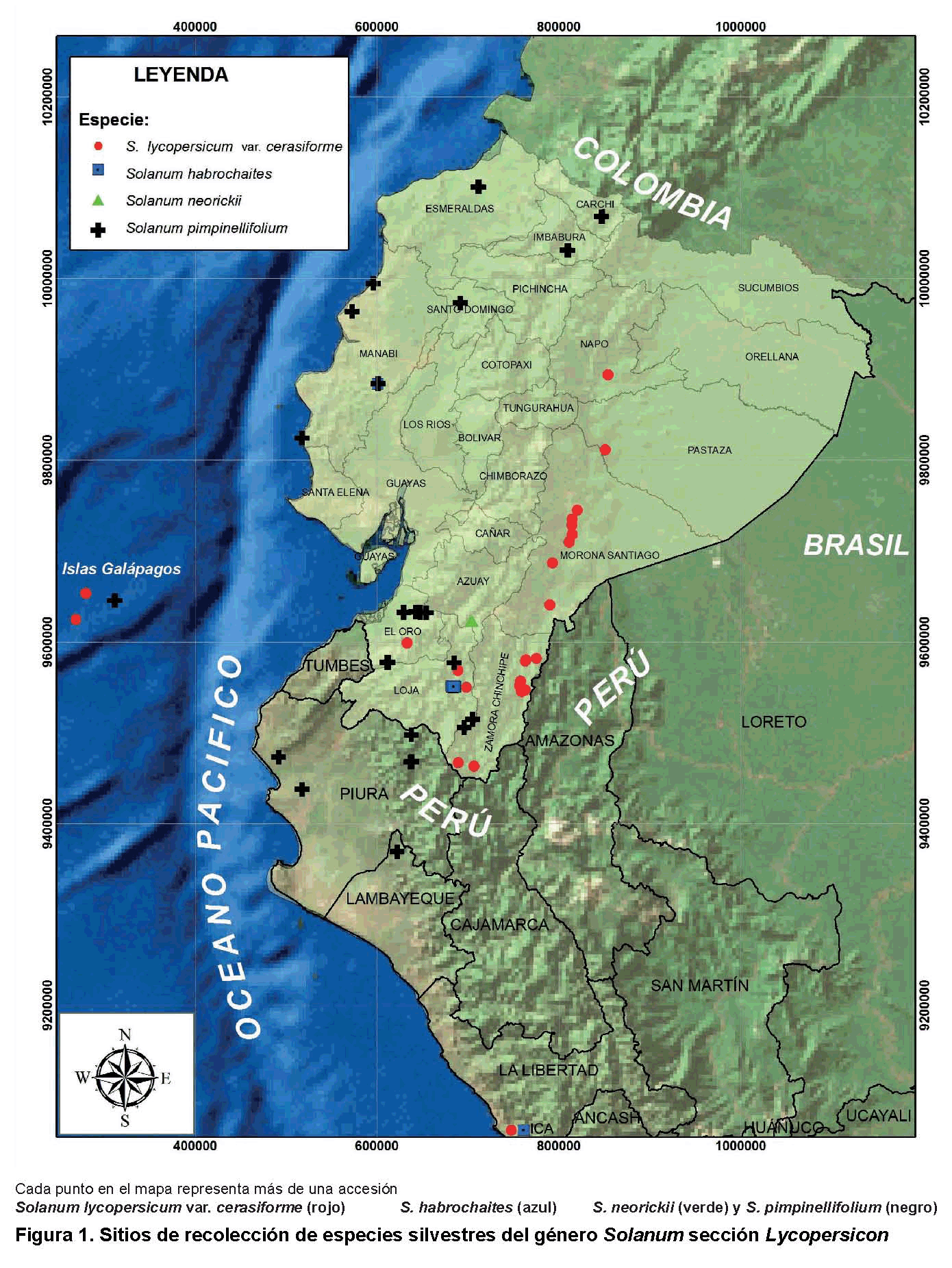
Las coordenadas geográficas se determinaron por un sistema global de posicionamiento basado en la ubicación relativa con respeto a un sistema artificial satélite (GPS, Magellan XL, San Dimas-California).
Se estudiaron cuatro especies silvestres del género Solanum sección Lycopersicon [dos del grupo Lycopersicon (Solanum pimpinellifolium L., Solanum lycopersicum var. cerasiforme Dunal), una del grupo Eriopersicon (Solanum habrochaites S. Knapp y D.M. Spooner) y una del grupo Arcanum (Solanum neorickii D.M. Spooner, G.J. Anderson y R.K. Jansen).
La caracterización morfológica se realizó en 659 plantas pertenecientes a 146 accesiones (en promedio cinco plantas por accesión) de las cuatro especies silvestres antes mencionadas (9), mientras que la caracterización molecular se estimó en 194 plantas, como se detalla en la Tabla I.

El análisis de las variables morfológicas se describe según diversos autores (9). En este estudio las variables no correlacionadas (≤ 0,85) se separaron en dos grupos: las vegetativas y las relacionadas con los componentes de la flor y el fruto. Con estos grupos se realizó un análisis de clasificación de coordenadas principales con las cuatro especies silvestres, utilizando el índice de similitud de Gower, mediante el paquete estadístico PAST (10).
Con estas mismas variables se obtuvo la matriz de distancia Euclidiana (cuantitativos) y de similitud (cualitativos), para construir un cladograma con el método de agrupamiento UPGMA. Posteriormente para comprobar la robustez de la construcción, se realizó una prueba de Mantel, mediante el programa NTSYSpc-2,02 g (11).
Para la caracterización molecular, el ADN fue extraído a partir de 100 mg de hojas conservadas a -86 °C, con el método del bromuro de cetil-trimetil amonio (CTAB) con algunas modificaciones . La calidad y cantidad del ADN extraído se estimó mediante electroforesis en geles de agarosa al 0,8 % y bromuro de etidio (3 %), corrido durante 50 minutos a 140 voltios en tampón TBE 1X. Las bandas se visualizaron en un transiluminador UVITEC BTS-20.LM a 320 nm y se comparó su intensidad con el patrón de bandas del marcador Trackit Lambda DNA/Hind III fragments (INVITROGEN). El ADN fue llevado a una concentración final de 10 ng/µL para un volumen final de 50 µL en una solución amortiguadora Tris EDTA.
De un total de 44 pares de cebadores de tipo microsatélite descritos (12), se escogieron 27, sobre la base de su polimorfismo y valoración de la calidad de las bandas (Tabla II).
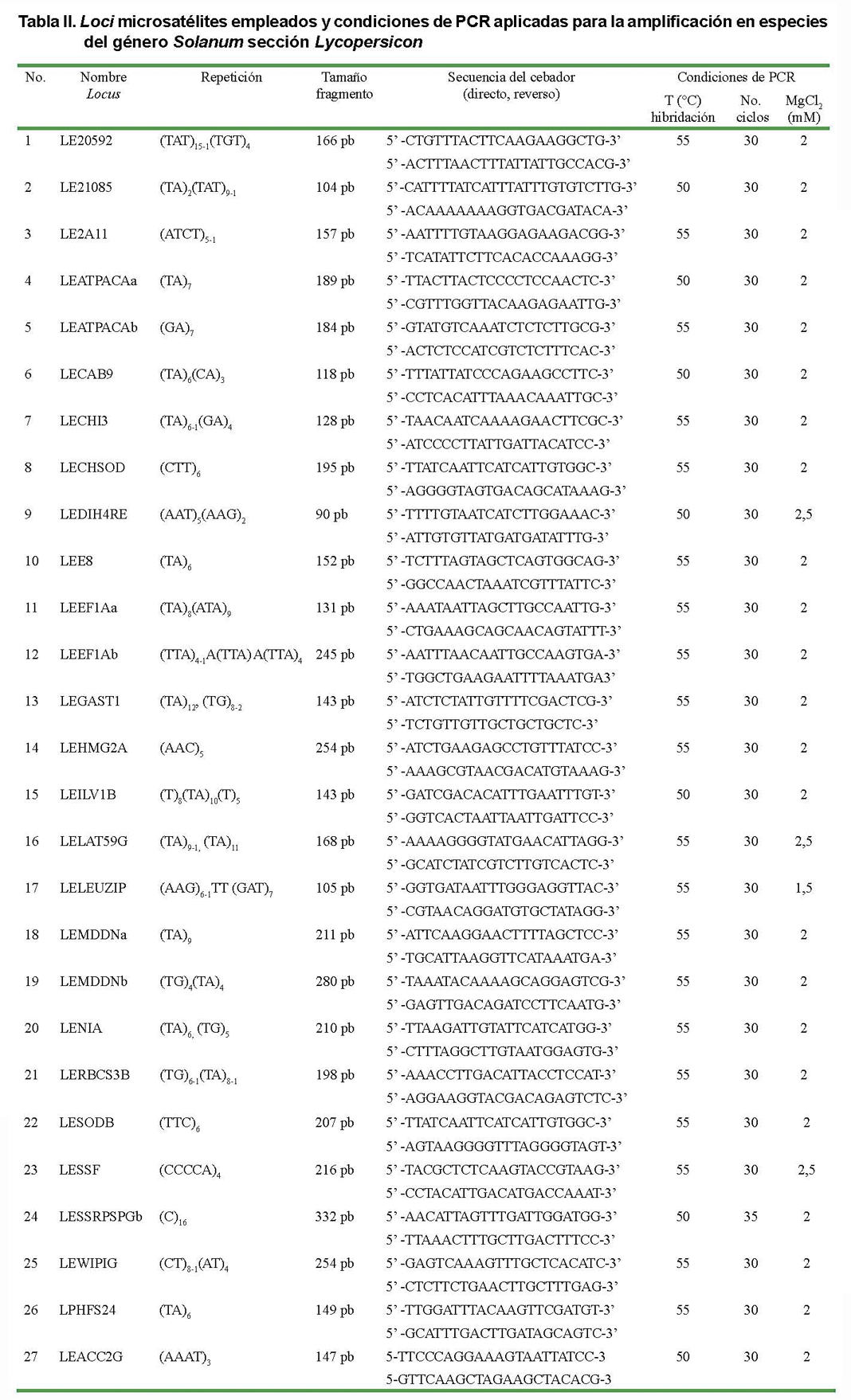
Cada reacción de amplificación se realizó para un volumen final de 15 µL: 1 µL de ADN genómico, 1,5 µL de 10X tampón [1X], 1,5 µL de cada cebador [1 µM], 1 µL de desoxirribonucleótidos [0,1mM] (BIOSYNTHESIS), 0,5 unidades de la enzima Taq ADN polimerasa (INVITROGEN) y MgCl2 hasta una concentración de 2 mM, con excepción de algunos loci en que se cambió la concentración final de la sal (Tabla II).
La PCR se realizó en un termociclador iCycler de BioRad, con el programa de amplificación descrito (12): un ciclo de 94 °C (3 min), 30 ciclos de la siguiente forma [55° (45 seg), 72° (1 min, 45 seg), 94 oC (45 seg)]. Un ciclo de extensión final a 55 °C (45 seg) y 72 °C (3 min). La temperatura de hibridación y el número de ciclos varió en dependencia de los cebadores empleados (Tabla II).
Los productos amplificados fueron separados mediante electroforesis vertical en geles desnaturalizantes PAGE, corridos a 75 W constantes, 45 a 50 oC, por 60 a 90 minutos, dependiendo del tamaño del fragmento esperado. Para asignar el tamaño a los alelos (pb), se empleó el marcador de tallas 30-330pb AFLP® DNA Ladder (LIFE TECHNOLOGIES).
El programa GenAlEx 6.1 (13) se empleó para estimar los valores medios de los parámetros de diversidad genética, teniendo en cuenta el tamaño de la muestra. El programa FSTAT versión 2.9.3 (14) fue usado para calcular índices de diversidad como: riqueza alélica (RA), heterocigosidad observada (Ho), diversidad intraespecífica (Hs), diversidad total (Ht), coeficiente de Nei de variación interespecífica (Dst), el estadígrafo FST de Weir y Cockerham y las diferencias estadísticas entre las especies, mediante la corrección de Bonferroni. El desequilibrio de ligamiento en las especies consideradas se llevó a cabo con 10 880 permutaciones y un ajuste del valor de probabilidad para el 5 % de 0,000092.
Se estimaron distancias genéticas individuales y con estas se construyeron árboles exploratorios mediante el algoritmo de agrupación Neighbor-Joining con los programas Population (15) y MEGA 5 (16).
En esta inferencia de la estructura genética se empleó también el programa Structure 2.2 (17). Este se corrió bajo las siguientes condiciones: 500 000 iteraciones para el período de presimulación y 1 000 000 de repeticiones de cadenas de Markov y Montecarlo (MCMC); se calcularon siete valores de k y se estimó el número de grupos (k) como el número maximizado al parámetro ∆k con Structure Harvester (18). El método utiliza un modelo con k poblaciones (demos) que asume están en equilibrio de Hardy-Weinberg (HW) y equilibrio gamético, aunque la primera premisa no se cumplió.
RESULTADOS Y DISCUSIÓN
El presente trabajo incluyó el estudio morfológico y molecular de cuatro especies silvestres del género Solanum recolectadas en su hábitat natural en territorio ecuatoriano. Las especies estudiadas han sido tradicionalmente subrepresentadas en estudios previos (19) y han mostrado diferencia en relación con las accesiones recolectadas en territorio peruano (20, 21). También se han caracterizado introducciones silvestres de tomate tipo cereza (S. lycopersicum) (21) para seleccionar genotipos sobre la base de caracteres de interés agronómico, para el mejoramiento del tomate cultivado.
Por otra parte, se han clonado recientemente genes que controlan la forma del fruto en S. lycopersicum (22). En los últimos dos años con la aplicación de la secuenciación de nueva generación, se refieren nuevos marcadores de los polimorfismos de nucleótido simple (SNPs, de sus siglas en inglés) que serán útiles en la caracterización de la estructura y diversidad genética de las especies silvestres y la búsqueda de loci ligados a caracteres cuantitativos (23, 24).
Caracterización morfológica
La Figura 2 muestra el resultado de los análisis de coordenadas principales, al considerar las variables morfológicas no correlacionadas en dos grupos (vegetativas y las relacionadas con la flor y el fruto). La especie S. habrochaites es la única que forma un conglomerado bien diferenciado, S. neorickii se diferencia respecto a S. habrochaites. La variedad cerasiforme no se diferencia por los caracteres empleados y aparece en todos los grupos.
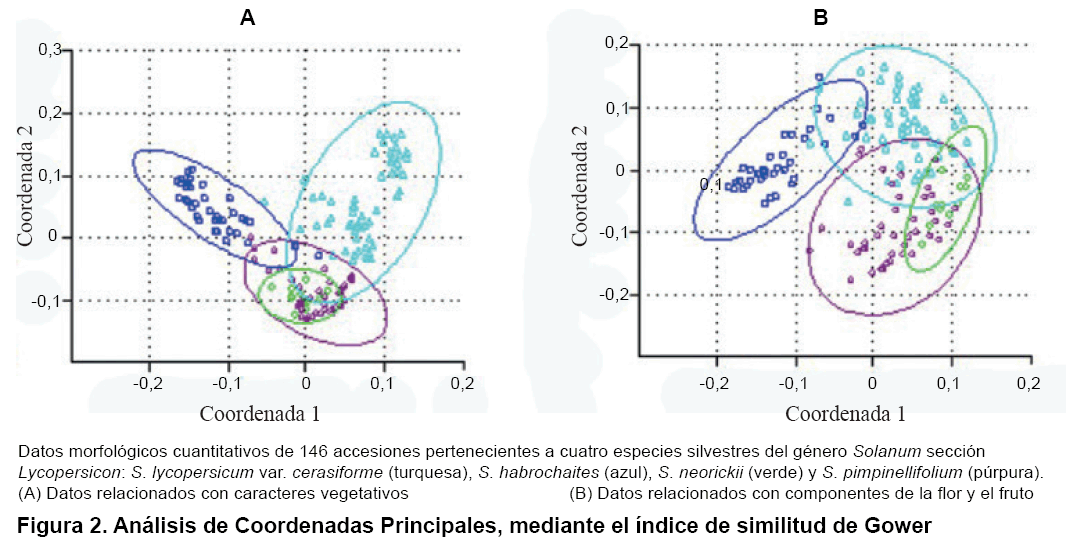
Los análisis de agrupamiento realizados (9), considerando juntas a todas las variables morfológicas no correlacionadas, fueron coincidentes con los del presente trabajo pues se formaron grupos con poca diferenciación de las especies, excepto S. habrochaites. De esta misma forma se comportó el análisis con las variables cualitativas.
Caracterización molecular
La caracterización molecular mediante 27 loci microsatélites mostró que diez de ellos no amplificaron con ninguna o algunas de las especies. Los cebadores LE21085 y LEGAST1 no amplificaron con S. lycopersicum var. cerasiforme; LEATPACAa, LEILV1B y LENIA con S. habrochaites; LESSF con S. pimpinellifolium; LE20592 y LELAT59G con S. neorickii y el cebador LEACC2G con ninguna especie; mientras que LESSRPSPGb amplificó únicamente con S. pimpinellifolium, por lo cual se lo considera potencialmente diagnóstico para la especie.
La caracterización molecular mediante 17 loci microsatélites que amplificaron con las cuatro especies, permitió obtener información sobre la estructura y diversidad genética de las especies silvestres estudiadas.
Estos 17 loci se encontraron en desequilibrio de ligamiento para el total de muestras y para cada especie por separado, lo que implica que cada locus brinda información independiente para determinar la estructura genética de las especies. Sin embargo, la mayor parte de las especies para estos loci mostraron un valor de probabilidad cercano al límite respecto a la falta de equilibrio genético de Hardy-Weinberg.
Este resultado se explica por el hecho de que cada accesión fue recolectada en sitios distantes (varios metros hasta decenas de kilómetros) a lo largo del territorio ecuatoriano y peruano y, por tanto, el conjunto de ellas no conforman una población. Otros autores encontraron falta de equilibrio genético en algunos loci microsatélites en las especies Solanum lycopersicoides y S. sitiens, cuya estructura y diversidad genética estudiaron (25).
Del mismo modo, hallaron altos coeficientes de consanguinidad, lo que argumentan planteando la existencia de efectos Walhund justificado por las áreas tan grandes a escala geográfica que están ocupadas por los grupos formados.
Los valores medios de los parámetros de variabilidad genética para los 17 loci analizados se muestran en la Tabla III. A pesar de que ninguno de los índices mostró diferencias significativas desde el punto de vista estadístico, debido a la gran varianza que presentan, la especie S. neorickii exhibió los menores valores.
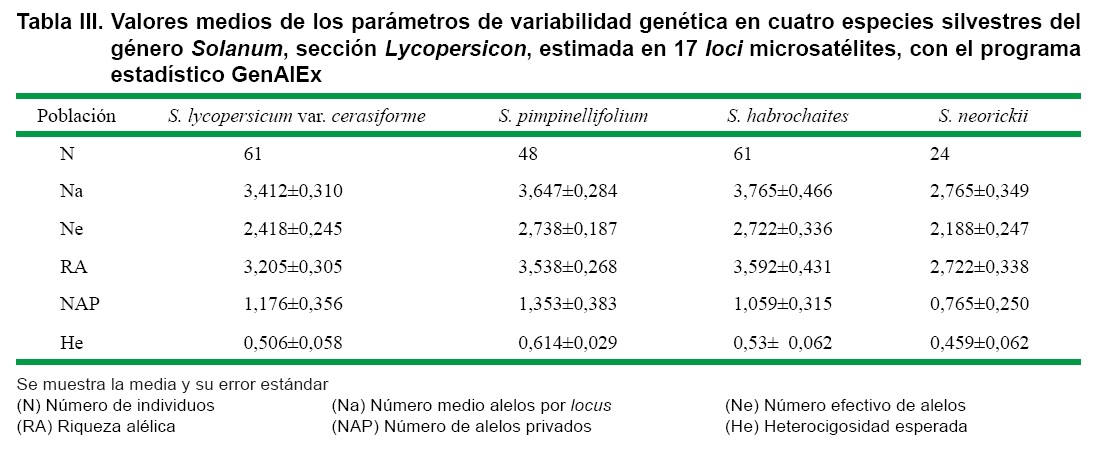
El hecho de que S. neorickii muestre valores más bajos en los índices de variabilidad genética, se explica debido a que la especie se encuentra en el Ecuador limitada en un pequeño espacio geográfico bajo las mismas condiciones climáticas, lo cual supone menor diversidad. S. pimpinellifolium con un tamaño de muestra inferior (22 %) respecto de S. lycopersicum var. cerasiforme muestra valores similares de variabilidad genética. Otros estudios refieren a S. pimpinellifolium como la especie con mayor variabilidad genética (20).
La especie está distribuida amplia y libremente a lo largo del borde de las carreteras desde territorio ecuatoriano hasta el Perú, lo cual ha permitido además obtener una amplia colección de la especie. A S. lycopersicum var. cerasiforme no es fácil hallarla y se encuentra restringida en los huertos de las casas de los aborígenes amazónicos. Se une a esto la dificultad para la alogamia debido al estigma inserto que presentan la mayoría de sus flores, todo lo cual justificaría una menor variación genética para esta especie, como lo obtenido en el presente trabajo.
En la medida en que la colección de S. habrochaites empleada en este estudio proviene de la zona de mayor concentración de la especie (Loja, al sur del Ecuador), se encontraron valores altos de variabilidad genética. Se une a la justificación de una mayor variabilidad, el hecho de que las poblaciones de la especie abundan a la orilla de los ríos, terrenos baldíos, laderas de montañas, jardines, entre otros.
Por otro lado, la condición de autoincompatibilidad que obliga a S. habrochaites a la polinización cruzada con individuos vecinos de la misma especie, puede justificar su menor variabilidad respecto a S. pimpinellifolium. Lo antes mencionado se apoya en lo encontrado por otros autores (19), quienes informan que las heterocigosidades observadas en una población del oeste de Loja-Ecuador son más bajas con respecto a las heterocigosidades que se esperarían en el supuesto de que la población se encontrase en equilibrio de Hardy-Weinberg, lo que significa que en todas las poblaciones existe cierto grado de autogamia o cruces entre plantas hermanas.
Con las distancias genéticas de Cavalli-Sforza de los individuos genotipados, se generó el árbol de distancias individuales que se muestra en la Figura 3 (A). En esta representación se observan cuatro grupos con límites bien marcados entre las especies: S. pimpinellifolium, S. habrochaites, S. neorickii y S. lycopersicum var. cerasiforme.
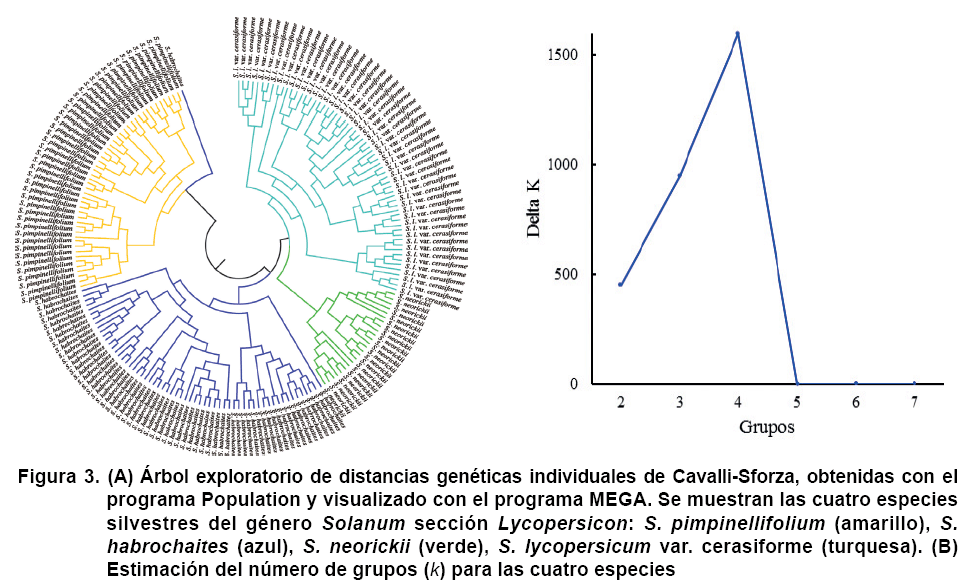
La estructura generada con la información de estos 17 loci, mediante el programa Structure, muestra un valor k de cuatro como el número maximizado al parámetro ∆k (Figura 3 B).
Los grupos formados por el programa Structure de la Figura 4 (B) muestran que S. lycopersicum var. cerasiforme (N=61) tiene coeficientes de asignación menores del 2 %, tanto de S. pimpinellifolium, S. neorickii, como de S. habrochaites.
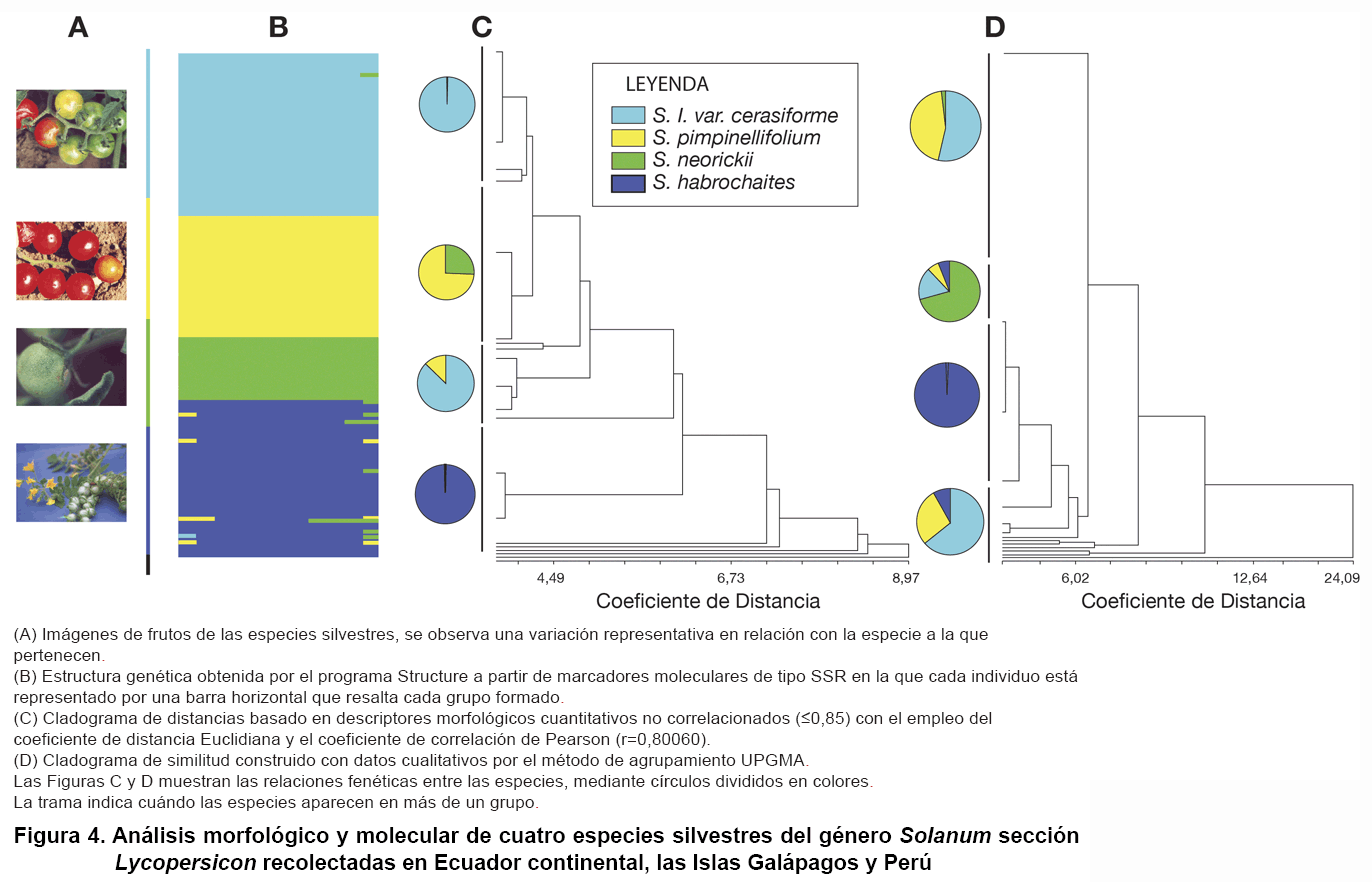
S. pimpinellifolium (N=48) constituye otro grupo, donde algunos individuos muestran porcentajes de asignación de S. neorickii (entre el 1 y el 5 %) y la mayoría muestran porcentajes de asignación de S. lycopersicum var. cerasiforme (entre 1 y 4 %). S. neorickii (N=24) muestra un pequeño coeficiente de asignación (1 %) de S. lycopersicum var. cerasiforme y S. habrochaites, mientras que S. habrochaites (N=61) presenta los mayores porcentajes de asignación de S. neorickii (entre 5 y 20 %) y de algunos individuos de S. lycopersicum var. cerasiforme y S. pimpinellifolium (1 %). Los valores de FST, mostraron congruencia total con la diferenciación de las especies descritas.
Debido a los bajos niveles de alogamia de S. lycopersicum var. cerasiforme, no es probable que exista un flujo genético entre esta especie y S. pimpinellifolium (20). También un trabajo con marcadores AFLP, entre estas dos especies refiere que son distintas genéticamente y existe poco flujo genético entre ellas (3). No obstante, S. lycopersicum var. cerasiforme presenta tanto plantas con flores de estigma inserto, como exerto y S. pimpinellifolium posee estigmas siempre exertos, lo cual podría favorecer el cruzamiento interespecífico y la alogamia entre las dos especies.
Los resultados producto de la caracterización fenotípica y genética de las cuatro especies silvestres propuestas, se muestran en la Figura 4. Los frutos de las cuatro especies silvestres estudiadas (Figura 4 A), presentan distinciones morfológicas que permiten diferenciarlas, no obstante el fruto no siempre está disponible.
La estructura genética de las especies silvestres (Figura 4 B), muestra a S. habrochaites y a S. neorickii diferenciadas cada una en un grupo individual, lo cual refleja que cada especie es producto de poblaciones con escasa rediferenciación a través del tiempo.
El cladograma obtenido mediante cortes arbitrarios de los nodos, a partir de las variables cuantitativas no correlacionadas (Figura 4 C), muestra a S. habrochaites en un grupo definido y separado de las especies restantes. S. pimpinellifolium y S. neorickii se encuentran mezcladas y separadas en otro grupo. Respecto a S. lycopersicum var. cerasiforme, aunque la mayor cantidad de individuos aparecen en un grupo definido, otros se encuentran junto a S. pimpinellifolium.
El cladograma construido con las variables cualitativas agrupadas por el método UPGMA que se presenta en la Figura 4 D, también separa a S. habrochaites de las otras especies; no obstante, las restantes aparecen mezcladas a lo largo del cladograma. El hecho de que S. pimpinellifolium esté junto con S. lycopersicum var. cerasiforme, se explica principalmente, debido a que las dos poseen frutos rojos comestibles.
CONCLUSIONES
- Las variables morfológicas son, en general, útiles en el reconocimiento de las especies silvestres del género Solanum sección Lycopersicon caracterizadas; sin embargo, permiten discriminar de manera efectiva a la especie Solanum habrochaites.
- Los loci microsatélites utilizados muestran una variabilidad genética similar entre las especies, con excepción de S. neorickii, con una variación genética menor. La estructura genética obtenida entre las especies, indica que estas se encuentran bien diferenciadas.
AGRADECIMIENTOS
A la Secretaria Nacional de Educación Superior, Ciencia y Tecnología del estado Ecuatoriano (SENESCYT), con cuyos fondos dotados a través de una beca, permitieron el financiamiento de la investigación.
BIBLIOGRAFÍA
1. Simbaqueba, J.; Cotes, A. M. y Barrero, L. S. ‘‘Linkage mapping of candidate genes for induce resistance and growth promotion by Trichoderma koningiopsis (Th003) in tomato Solanum lycopersicum’’. Acta Biológica Colombiana, vol. 16, no. 2, agosto de 2011, pp. 47-62, ISSN 0120-548X.
2. Lucatti, A. F.; van Heusden, A. W.; de Vos, R. C.; Visser, R. G. y Vosman, B. ‘‘Differences in insect resistance between tomato species endemic to the Galapagos Islands’’. BMC Evolutionary Biology, vol. 13, 2013, p. 175, ISSN 1471-2148, DOI 10.1186/1471-2148-13-175.
3. Nakazato, T. y Housworth, E. A. ‘‘Spatial genetics of wild tomato species reveals roles of the Andean geography on demographic history’’. American Journal of Botany, vol. 98, no. 1, 1 de enero de 2011, pp. 88-98, ISSN 0002-9122, 1537-2197, DOI 10.3732/ajb.1000272.
4. Peralta, I. E.; Knapp, S. y Spooner, D. M. ‘‘Nomenclature for wild and cultivated tomatoes’’. Tomato Genetics Cooperative Report, vol. 56, 2006, pp. 6–12.
5. Koenig, D.; Jiménez, J. M.; Kimura, S.; Fulop, D.; Chitwood, D. H.; Headland, L. R.; Kumar, R.; Covington, M. F.; Devisetty, U. K.; Tat, A. V.; Tohge, T.; Bolger, A.; Schneeberger, K.; Ossowski, S.; Lanz, C.; Xiong, G.; Taylor, T. M.; Brady, S. M.; Pauly, M.; Weigel, D.; Usadel, B.; Fernie, A. R.; Peng, J.; Sinha, N. R. y Maloof, J. N. ‘‘Comparative transcriptomics reveals patterns of selection in domesticated and wild tomato’’. Proceedings of the National Academy of Sciences, vol. 110, no. 28, 7 de septiembre de 2013, pp. 2655-2662, ISSN 0027-8424, 1091-6490, DOI 10.1073/pnas.1309606110.
6. Geethanjali, S.; Kadirvel, P.; Peña, R. de la; Rao, E. S. y Wang, J. F. ‘‘Development of tomato SSR markers from anchored BAC clones of chromosome 12 and their application for genetic diversity analysis and linkage mapping’’. Euphytica, vol. 178, no. 2, 25 de diciembre de 2010, pp. 283-295, ISSN 0014-2336, 1573-5060, DOI 10.1007/s10681-010-0331-8.
7. Martins, K.; Chaves, L. J.; Vencovsky, R. y Kageyama, P. Y. ‘‘Genetic structure based on nuclear and chloroplast microsatellite loci of Solanum lycocarpum A. St. Hil. (Solanaceae) in Central Brazil’’. Genetics and Molecular Research, vol. 10, no. 2, 2011, pp. 665–677, ISSN 1676-5680, DOI 10.4238/vol10-2gmr1046.
8. El-Awady, M. A.; El-Tarras, A. A. y Hassan, M. M. ‘‘Genetic diversity and DNA fingerprint study in tomato (Solanum lycopersicum L) cultivars grown in Egypt using simple sequence repeats (SSR) markers’’. African Journal of Biotechnology, vol. 11, no. 96, 2012, p. 16233, ISSN 1684–5315, DOI 10.5897/AJB12.2477.
9. Morales, P. M. N.; Espinosa, L. G.; Morales, A. Á. R.; Sánchez, M. B. R.; Jiménez, C. Á. M. y Milián, G. Y. ‘‘Caracterización morfológica y evaluación de resistencia a Fusarium oxysporum en especies silvestres del género Solanum sección Lycopersicon’’. Revista Colombiana de Biotecnología, vol. 16, no. 1, 2014, pp. 62–73, ISSN 1909-8758.
10. Hammer, O. D.; Harper, D. A. T. y Ryan, P. D. ‘‘PAST-Paleontological Statistics software package for education and data analysis’’. Palaeontología Electrónica, vol. 4, no. 1, 2007, pp. 1-9, ISSN 1094-8074.
11. Rohlf, F. J. Ntsys-pc: Numerical taxonomy and multivariate analysis system: the principles and practice of numerical classification. versión 2.02, edit. Exeter software, Setauket-New York, 1998, ISBN 0-925031-30-5.
12. Smulders, M. J. M.; Bredemeijer, G.; Rus-Kortekaas, W.; Arens, P. y Vosman, B. ‘‘Use of short microsatellites from database sequences to generate polymorphisms among Lycopersicon esculentum cultivars and accessions of other Lycopersicon species’’. Theoretical and Applied Genetics, vol. 94, no. 2, febrero de 1997, pp. 264-272, ISSN 0040-5752, 1432-2242, DOI 10.1007/s001220050409.
13. Peakall, R. y Smouse, P. E. ‘‘GenAlEx 6: genetic analysis in Excel. Population genetic software for teaching and research’’. Molecular Ecology Notes, vol. 6, no. 1, 1 de marzo de 2006, pp. 288-295, ISSN 1471-8286, DOI 10.1111/j.1471-8286.2005.01155.x.
14. Goudet, J. FSTAT, a program to estimate and test gene diversities ad fixation indices [en línea]. versión 2.9.3, edit. Institute of Ecology, Lausanne, Switzerland, 2001, [Consultado: 25 de marzo de 2013], Disponible en: <http://www2.unil.ch/popgen/softwares/fstat.html>.
Langella, O. Populations. Logiciel de génétique des populations [en línea]. versión 1.2.32, 2002, [Consultado: 19 de mayo de 2012], Disponible en: <http://www.bioinformatics.org/project/?group_id=84>.
15. Tamura, K.; Peterson, D.; Peterson, N.; Stecher, G.; Nei, M. y Kumar, S. ‘‘MEGA5: Molecular Evolutionary Genetics Analysis Using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods’’. Molecular Biology and Evolution, vol. 28, no. 10, 10 de enero de 2011, pp. 2731-2739, ISSN 0737-4038, 1537-1719, DOI 10.1093/molbev/msr121.
16. Pritchard, J. K.; Stephens, M. y Donnelly, P. ‘‘Inference of Population Structure Using Multilocus Genotype Data’’. Genetics, vol. 155, no. 2, 1 de junio de 2000, pp. 945-959, ISSN 0016-6731, 1943-2631.
17. Earl, D. A. y vonHoldt, B. M. ‘‘STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method’’. Conservation Genetics Resources, vol. 4, no. 2, 13 de octubre de 2011, pp. 359-361, ISSN 1877-7252, 1877-7260, DOI 10.1007/s12686-011-9548-7.
18. Sifres, A.; Blanca, J. y Nuez, F. ‘‘Pattern of genetic variability of Solanum habrochaites in its natural area of distribution’’. Genetic Resources and Crop Evolution, vol. 58, no. 3, 11 de junio de 2010, pp. 347-360, ISSN 0925-9864, 1573-5109, DOI 10.1007/s10722-010-9578-0.
19. Zuriaga, E.; Blanca, J. M.; Cordero, L.; Sifres, A.; Blas-Cerdán, W. G.; Morales, R. y Nuez, F. ‘‘Genetic and bioclimatic variation in Solanum pimpinellifolium’’. Genetic Resources and Crop Evolution, vol. 56, no. 1, 16 de mayo de 2008, pp. 39-51, ISSN 0925-9864, 1573-5109, DOI 10.1007/s10722-008-9340-z.
20. Agudelo, A. G.; Ceballos, N. y Orozco, F. J. ‘‘Caracterización morfológica del tomate tipo cereza (Solanum lycopersicum Linnaeus)’’. Agronomía, vol. 19, no. 2, 2011, pp. 44-53, ISSN 0568-3076.
21. Rodríguez, G. R.; Muños, S.; Anderson, C.; Sim, S. C.; Michel, A.; Causse, M.; Gardener, B. B. M.; Francis, D. y van der Knaap, E. ‘‘Distribution of SUN, OVATE, LC, and FAS in the Tomato Germplasm and the Relationship to Fruit Shape Diversity’’. Plant Physiology, vol. 156, no. 1, 1 de mayo de 2011, pp. 275-285, ISSN 1532-2548, DOI 10.1104/pp.110.167577.
22. Víquez, Z. M.; Vosman, B.; van de Geest, H.; Bovy, A.; Visser, R. G.; Finkers, R. y van Heusden, A. W. ‘‘Tomato breeding in the genomics era: insights from a SNP array’’. BMC Genomics, vol. 14, no. 1, 2013, p. 354, ISSN 1471-2164, DOI 10.1186/1471-2164-14-354.
23. Lin, T.; Zhu, G.; Zhang, J.; Xu, X.; Yu, Q.; Zheng, Z.; Zhang, Z.; Lun, Y.; Li, S.; Wang, X.; Huang, Z.; Li, J.; Zhang, C.; Wang, T.; Zhang, Y.; Wang, A.; Zhang, Y.; Lin, K.; Li, C.; Xiong, G.; Xue, Y.; Mazzucato, A.; Causse, M.; Fei, Z.; Giovannoni, J. J.; Chetelat, R. T.; Zamir, D.; Städler, T.; Li, J.; Ye, Z.; Du, Y. y Huang, S. ‘‘Genomic analyses provide insights into the history of tomato breeding’’. Nature Genetics, vol. 46, no. 11, noviembre de 2014, pp. 1220-1226, ISSN 1061-4036, DOI 10.1038/ng.3117.
24. Albrecht, E.; Escobar, M. y Chetelat, R. T. ‘‘Genetic diversity and population structure in the tomato-like nightshades Solanum lycopersicoides and S. sitiens’’. Annals of Botany, vol. 105, no. 4, 4 de enero de 2010, pp. 535-554, ISSN 0305-7364, 1095-8290, DOI 10.1093/aob/mcq009.
Recibido: 13/04/2015
Aceptado: 02/11/2015
M.Cs. María N. Morales Palacio, IDENTIGEN. Laboratorio Biomolecular, Consorcio Torre 1, Azuay y Sucre, Loja, Ecuador. Email: nataliamorales88@yahoo.es






