
ARTÍCULO ORIGINAL
Caracterización de rizobios aislados de nódulos de frijol caupí, en suelos salinos de Cuba
Characterization of rhizobia isolated from cowpea nodules, in Cuban saline soils
Ernesto Gómez-Padilla,I Beatriz Ruiz-Díez,II Susana Fajardo,II Bettina Eichler-Loebermann,III Roeland Samson,IV Patrick Van-Damme,V Raúl López-Sánchez,I Mercedes Fernández-PascualII
ICentro de Estudio de biotecnología Vegetal, Facultad de Ciencias Agrícolas, Universidad de Granma, Cuba.
IIInstituto de Ciencias Agrícolas, ICA-CSIC, Madrid, Spain.
IIIUniversidad de Rostock, Facultad de Ciencias Agrícolas y Ambientales, Rostock, Alemania.
IVUniversidad de Antwerp, Facultad de Ciencias de la Ingeniería de Biociences. Belgium.
VUniversidad de Ghent, Laboratorio de Agronomía Tropical y Subtropical y Etnobotánica, Belgium.
RESUMEN
A partir de suelos del Valle del Río Cauto (Granma, Cuba) afectados por sales, se aislaron seis bacterias procedentes de nódulos de Vigna unguiculata (L.) Walp. Los aislados se sometieron a diferentes niveles de sales (0,17 - 6,6 dS m-1 de NaCl), pH (4,5 - 9) y temperatura (28 - 45 oC), con el objetivo de determinar su tolerancia a estos tipos de estrés. La variación en el gen 16S rRNA, se examinó a través del análisis de restricción de amplificados 16S rDNA (ARDRA) y secuenciación directa. Tres aislados, VIBA-1, VIBA-2 y VIBA-6, mostraron resultados similares al control (0,17 dS m-1) con 2,6 y 3,4 dS m-1 de NaCl. Todas las cepas crecieron a pH 7 y 9 hasta 40 oC, solo dos de ellas (VIBA-4 y VIBA-5) crecieron a 45 oC. El análisis de filogenia, confirmó la estrecha relación de la cepa Bradyrhizobium liaoningense con el aislamiento VIBA-1, y los aislamientos VIBA-2, VIBA-3, VIBA-5 y VIBA-6, con la cepa Bradyrhizobium yuanmingense, y uno de ellos a Rhizobium radiobacter (formalmente Agrobacterium tumefaciens, VIBA-4). Todos los aislados con excepción de VIBA-4, formaron nódulos en las plantas cuando fueron inoculadas. La sobrevivencia de estas cepas en condiciones de estrés (salinidad, alcalinidad y temperatura), evidencia su habilidad para crecer bajo estas condiciones ambientales específicas. Los resultados indicaron que, en un entorno de suelos salinos de Cuba, existen cepas de Bradyrhizobium que pueden establecer simbiosis con Caupí, pero la diversidad de estos microorganismos es aún escasa, debido a que sólo dos especies lograron nodular la leguminosa.
Palabras clave: Bradyrhizobium sp, identificación molecular, nodulación, salinidad, tolerancia, Vigna unguiculata.
ABSTRACT
In soils of Cauto River Valley (Granma, Cuba) affected by salts, six new bacteria isolated from nodules of Vigna unguiculata (L.) Walp were obtained. The six isolates were subjected to differents salt levels (0,17- 6,6 dS m-1 of NaCl), pH levels (4,5 - 9) and temperatures (28 - 45 oC) with the objective to determine their tolerance to these abiotic stresses. Variation within the 16S rRNA gene, was examined by amplified 16S rDNA restriction analysis (ARDRA) and direct sequencing to show genetic diversity. Three isolates (VIBA-1, VIBA-2 and VIBA-6) achieved similar results as the control with 2,6 and 3,4 dS m-1 of NaCl. All of the isolates could grow at pH 7 and 9 and got growth until 40 oC, only two of them (VIBA-4 and VIBA-5) grew at 45 oC. Phylogeny analysis confirmed the close relationship of one isolate with Bradyrhizobium liaoningense (VIBA-1) and four isolates with Bradyrhizobium yuanmingense (VIBA-2, VIBA-3, VIBA-5 and VIBA-6) and one to Rhizobium radiobacter (formely Agrobacterium tumefaciens, VIBA-4). All of them, with the exception of VIBA-4, were able to nodulate in the plants when they were inoculated. The survival of these strains in the different abiotic stress (salinity, alkalinity and temperature), evidenced their ability to grow under this specific environment. The findings indicated that under Cuban saline soils there are Bradyrhizobium strains able to establish symbiosis with cowpea, but the diversity is still low, being characterized only two different species able to nodulate.
Key words: Bradyrhizobium sp, molecular identification, nodulation, salinity, tolerance, Vigna unguiculata.
INTRODUCCIÓN
La salinidad es uno de los principales problemas en los ecosistemas agrícolas en el mundo; cerca del 50 % del planeta está afectado por este flagelo (1,2). Cuba no es la excepción, ya que se han informado alrededor de un millón de hectáreas de suelos salinos. El valle del río Cauto, ubicado en la región oriental del país, tiene 228 mil hectáreas afectadas por la salinidad; entre ellos, el 28 % están clasificados como fuertemente salinos y el 11 % muy fuertemente salinos (3). Se han producido numerosos daños en el rendimiento de los cultivos, incluso algunas áreas de cultivo han sido abandonadas, causadas por la pérdida de capacidad de las especies de plantas para crecer en estos ambientes salinos (3).
Por otro lado, se sabe que la salinidad no solo perjudica fisiológica y bioquímicamente a las plantas. De hecho, las comunidades microbianas, entre ellas las bacterias rizobios, disminuyen en número, diversidad y actividad, cuando los suelos dejan de cultivarse y cuando se han encontrado las condiciones de estrés ambiental, como salinidad del suelo, temperatura, pH, metales pesados (4,5).
En esta situación, una opción natural y ecológica para disminuir el efecto nocivo de las sales, y para mantener la producción bajo estrés salino, puede ser la selección e introducción de plantas o microorganismos adaptados a estas condiciones (6).
En este sentido, el frijol caupí es una opción debido a que es reportada como especie rústica que puede ser encontrada en ambientes adversos y con tolerancia a la sequía, alta temperatura, metales pesados y estrés salino, pero también puede contribuir con una cantidad apreciable de nitrógeno (150 kg N ha-1) de fijación simbiótica al ecosistema (7,8). Sin embargo, el aislamiento de cepas rizobiales nativas adaptadas a estreses abióticos específicos y capaces de establecer una simbiosis eficiente con el caupí es una práctica obligatoria en las áreas salinas de la provincia Granma (Valle del Río Cauto) Cuba, ya que no existen cepas comerciales para cubrir la inoculación de Vigna para estos suelos. Además, la inoculación de semillas de caupí y plántulas con bradyrhizobios nativos apropiados, puede garantizar la nodulación de la raíz y el aumento de la producción de la planta (9).
Este estudio se llevó a cabo con el objetivo de aislar bacterias rizobiales nativas de suelos salinos, evaluar la tolerancia al estrés abiótico (solución salina, pH y alta temperatura), realizar la identificación genética y obtener una colección de bioinoculantes de caupí en suelos salinos del valle del río Cauto en Cuba.
MATERIALES Y MÉTODOS
AISLAMIENTO BACTERIANO
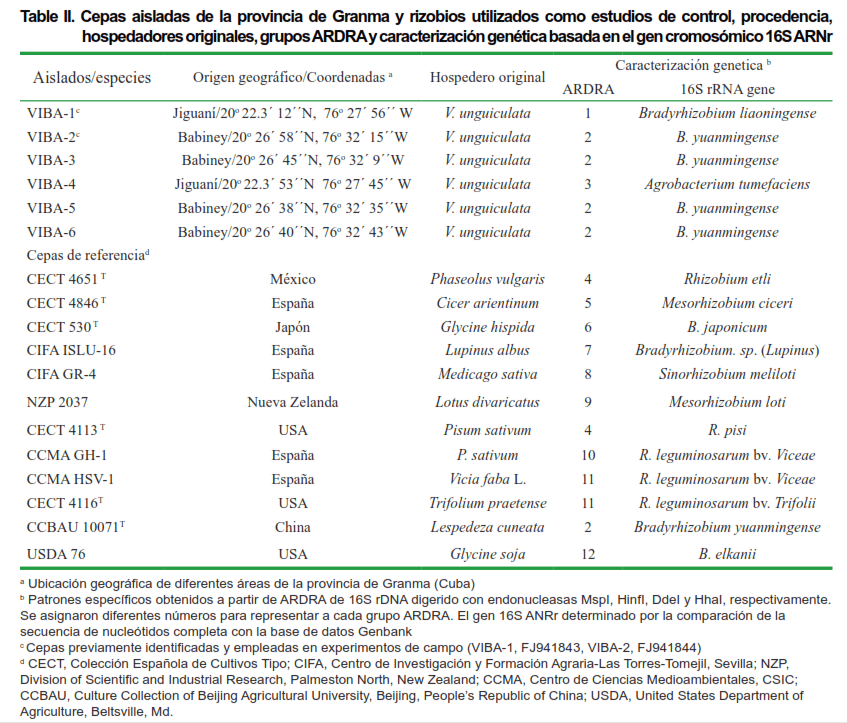
Los aislados utilizados en este estudio, se obtuvieron a partir de nódulos de raíces de variedades de Caupí [Vigna unguiculata (L.) Walp.], cultivadas en suelos salinos en los municipios de Babiney (con 4,76 dS m-1) de conductividad eléctrica (CE) y Jiguaní (5,8 dS m-1 EC), dos localidades del Valle del Río Cauto (Provincia de Granma, Cuba). Se utilizaron 12 cepas de referencia de la colección internacional como patrón de control. Los aislados, los sitios de origen y las cepas de referencia se describen en la Tabla I. El aislamiento de bacterias se realizó a partir de nódulos recuperados directamente de las variedades de caupí establecidas en áreas experimentales. Los nódulos se desinfectaron superficialmente con etanol (95 % v/v) durante 30 s, luego se sometieron a HgCl2 0,1 % (p/v) durante 45 s, y se lavaron con agua destilada estéril. Los nódulos se cortaron y se transfirió un asa llena de células infectadas al medio sólido de extracto de levadura (YEM) (10). Se obtuvieron colonias individuales y se verificó su pureza mediante rayas repetidas y examen microscópico. Los aislados se cultivaron rutinariamente a 28 ºC en medio líquido YEM (10). Las cepas de referencia de colecciones internacionales se usaron como patrón de control (Tabla II).
CARACTERIZACIÓN FENOTÍPICA
Para evaluar su capacidad de tolerancia al estrés abiótico las cepas VIBA-1, VIBA-2, VIBA-3, VIBA-4, VIBA-5 y VIBA-6 y las cepas de referencia CCBAU 10071 (Bradyrhizobium yuanmingense) se sometieron a sal, pH y temperaturas extremas (11).
Se usó un cultivo estándar para llevar a cabo los experimentos (50 μl de cultivo en fase exponencial sobre 5 ml de cultivo final, dilución 1:100). Se colocaron 5 ml de medio YEM líquido estéril en tubos de vidrio Vimex esterilizados.
Cada cepa se sembró en condiciones estériles y luego, se colocó en una incubadora a 28 oC y 100 rpm hasta alcanzar el crecimiento exponencial.
TOLERANCIA AL ESTRÉS SALINO
Cada aislamiento se sometió a 0,17; 2,6; 3,4; 4,2; 5,00; 5,8 y 6,6 dS m-1 de NaCl respectivamente y luego se colocaron en tres tubos de vidrio. El nivel de NaCl recomendado para los cultivos de Rhizobium (0,1 g L-1 = 1,7 mM = 0,17 dS m-1 se consideró como control (10). El crecimiento microbiano de cada tratamiento se determinó a 120 h después de sembrar los cultivos (fase estacionaria) a través de la densidad óptica (medida de absorbancia a 680 nm) utilizando un espectrofotómetro Spectronic 2000 (Bausch & Lomb).
TOLERANCIA AL ESTRÉS DE pH
Los aislados se sometieron a tres tratamientos de medio YEM de cultivo regulado a pH 4,5, 7 y 9. Se usaron ácido 2,2-dimetilsuccínico (30 mM) y clorhidrato de tris (50 mM) para obtener soluciones a pH 4,5 y pH 9, respectivamente. Los pH se verificaron después del tratamiento en autoclave y no cambiaron en más de ± 0,1 unidades. Se siguió el crecimiento microbiano de cada tratamiento como en los ensayos de salinidad.
TOLERANCIA AL ESTRÉS DE TEMPERATURA
Los aislados se cultivaron en placas de Petri en medio sólido YEM (10, y se sometieron a 28 (control), 37, 40, 45 oC, en los que se observó su crecimiento cada 24 horas durante 12 días (11).
PRUEBA DE NODULACIÓN
Se llevó a cabo una prueba de inoculación para evaluar la capacidad de los aislados para formar nódulos de raíces (10). Las plantas de Caupí se inocularon con todas las cepas nativas, se utilizó un control sin inoculación. Las plantas de semillero se cultivaron en una cámara de crecimiento bajo un fotoperíodo de 14 h de luz (23 ºC)/10 h de oscuridad (20 ºC) durante un mes. Las plantas se regaron con agua destilada esterilizada durante la primera semana después de la siembra y con solución nutritiva sin nitrógeno el resto del tiempo.
CARACTERIZACIÓN MOLECULAR
Aislamiento del DNA genómico
Se empleó una colección de cepas de rizobios conocidas como referencia (Tabla II).
Se usó un asa de cada aislado para inocular 20 ml
de caldo YEM. Después de la incubación (10), las células se recogieron, se lavaron dos veces con solución salina tamponada con fosfato (PBS) y se aisló el ADN usando el kit de aislamiento de ADN microbiano UltraClean TM (MOBIO). La concentración e integridad del ADN se analizaron mediante electroforesis con geles de agarosa al 0,8 % y se compararon con cantidades conocidas de ADN del fago lambda (12).
AMPLIFICACIÓN POR PCR DE 16S ADNr
El gen 16S ADNr de todas las cepas se amplificó por PCR. Los cebadores fD1 y rP2 se emplearon a una concentración final de 1 μM y se sintetizaron por Genotek (España). La amplificación se desarrolló utilizando un termociclador (Veriti 96 Well, Applied Biosystems) (13).
ANÁLISIS DE RESTRICCIÓN (ARDRA)
Las endonucleasas de restricción MspI, HinfI, DdeI y HhaI (New England Biolabs) se usaron en reacciones de digestión separadas con ADNr 16S amplificado por PCR de todos los aislados enumerados en la Tabla II. Se digirió una porción de 15 μl de reacciones de amplificación de acuerdo con las recomendaciones del fabricante y los análisis de restricción del ADNr 16S amplificado (ARDRA) (13).
SECUENCIA DE ADNr 16S
Las secuencias de productos de PCR se obtuvieron con un secuenciador ABI PRISM 3700 (Applied Biosystems) usando los sistemas de ciclo Taq Dyedeoxi Terminator (Automatic Sequencing Service SAAD, CIB, CSIC, Madrid). Se usaron 14 μl de producto de PCR para reacciones de secuenciación y 1,4 μl de los cebadores 1050R, 800R, 800F e IRF1 a 5 μM (12). La secuencia de cada aislado se determinó mediante alineamientos por pares utilizando el análisis Clustal W. Las secuencias de similitudes fueron identificadas y analizadas por el programa BLASTN en el servicio de red del GeneBank (http://blast.ncbi.nlm.nih.gov/Blast.cgi). Los valores de porcentaje de similitud con respecto al gen 16S rRNA se calcularon mediante alineaciones múltiples de Clustal W (http://www.npsa-pbil.ibcp.fr/) (13).
ANÁLISIS FILOGENÉTICO
Los análisis filogenéticos se realizaron utilizando el programa MEGA versión 4 (14). El árbol filogenético se construyó utilizando el método de unión de vecinos basado en el modelo de distancia de dos parámetros de Kimura (15). Para evaluar el soporte relativo para cada grupo taxonómico, los valores de arranque (bootstrap) se calcularon a partir de 1,000 análisis replicados.
DISEÑO EXPERIMENTAL Y ANÁLISIS ESTADÍSTICO
Los experimentos se organizaron en diseños completamente aleatorizados y se repitieron al menos dos veces, con tres repeticiones cada vez. Los datos de tolerancia al estrés salino y a los experimentos de estrés por pH se analizaron mediante un análisis de varianza simple (p<0,05). Las medias de cada tratamiento se compararon usando la prueba de Tukey (16). Todos los análisis estadísticos se realizaron con Statistica para Windows, versión 10 (17).
RESULTADOS
CARACTERIZACIÓN FENOTÍPICA
Todos los aislados fueron Gram negativos y se produjo una cantidad considerable de polisacáridos en VIBA-2.
El período de crecimiento de las cepas fue diferente, VIBA-1 comenzó a crecer al quinto día después de la siembra, y por lo tanto tomó el período de tiempo más largo. El resto de las cepas mostraron un crecimiento intermedio en el rango de tres o cuatro días (datos no mostrados).
TOLERANCIA AL ESTRÉS SALINO
El aumento de la concentración de salinidad en el medio de cultivo indujo una reducción significativa del crecimiento en las cepas, particularmente para niveles superiores a 4,2 dS m-1, donde los valores de absorbancia disminuyeron con el aumento de la concentración de sal (Figura 1). Los aislados VIBA-1 y VIBA-2 mostraron el mayor crecimiento en el rango de 2,6 a 3,4 dS m-1 de NaCl, con resultados similares al control. El crecimiento de todas las cepas se redujo significativamente con 4,2 dS m-1 de NaCl.
Por otro lado, CCBAU 10071 (Bradyrhizobium yuanmingense) reveló un mayor crecimiento que el nivel de salinidad de control (0,17 dS m-1) a 2,6 dS m-1 de NaCl. Incluso esta cepa alcanzó un crecimiento similar al control con los niveles más altos de sal (6,6 dS m-1) (Figura 1). Esta cepa podría considerarse la más tolerante al estrés salino.
TOLERANCIA AL ESTRÉS DE pH
Ninguna de las cepas pudo crecer a pH 4,5. El crecimiento de los aislados, VIBA-1, VIBA-2, VIBA-3 y VIBA-6 aumentó significativamente a pH 7 en comparación con pH 9. Sin embargo, VIBA-4, VIBA-5 y la cepa de referencia CCBAU 10071 fueron similares para ambos niveles de pH, incluso este último, tuvo la tendencia a crecer mejor en pH 9 (Figura 2).
TOLERANCIA AL ESTRÉS DE TEMPERATURA
Todos los aislamientos, incluida la cepa de referencia, podrían crecer hasta 40 oC a la misma velocidad que 28 ºC (Tabla I). Las excepciones fueron VIBA-4 y VIBA-5, que crecieron a 45 oC aunque a menor velocidad. Estos resultados sugieren una muy buena adaptación de estos aislamientos a las altas temperaturas que son características de las áreas donde se aislaron las bacterias.
PRUEBA DE NODULACIÓN
La inoculación de las plantas con VIBA-1, VIBA-2, VIBA-3, VIBA-5 y VIBA-6, indujó la formación de nódulos lo que indica que estas cepas pertenecen a la familia Rhizobiaceae. Además, el color rosado dentro de los nódulos reveló su actividad de fijación de nitrógeno, debido a la presencia de leghemoglobina en la zona infectada (18). Sin embargo, VIBA-4 no fue capaz de inducir la nodulación en las tres ocasiones en que se realizó el experimento.
CARACTERIZACIÓN MOLECULAR
Análisis de restricción del ADNr 16S amplificado (ARDRA)
El 16S ADNr de cada aislado y las cepas de referencia se amplificaron produciendo una banda característica de 1,500 pb. El análisis de restricción mostró de cuatro a ocho patrones para cada enzima utilizada (no se muestran los patrones de restricción). Se obtuvieron doce genotipos ARDRA (1-12), que representan las diferentes combinaciones de patrones de restricción, de todas las cepas investigadas (Tabla II).
TIPO DE CEPA
Los seis aislados de caupí se clasificaron en tres genotipos diferentes de ADNr. VIBA-2, VIBA-3, VIBA-5 y VIBA-6 exhibieron el mismo patrón de genotipo que Bradyrhizobium yuanmingense CCBAU 10071 (Tabla II), mientras que los genotipos de VIBA-1 y VIBA-4 no coincidieron con ninguna cepa de referencia. Utilizando los resultados de referencia ARDRA, se realizó la determinación de las secuencias del gen 16S ADNr para lograr la afiliación de la especie y su identidad genética.
16S ANÁLISIS DE SECUENCIA DE ADNr
Las secuencias se introdujeron en la base de datos GeneBank con los siguientes números de acceso: FJ941843, VIBA-1; FJ941844, VIBA-2; FJ941845, VIBA-3; GU784791, VIBA-4; GU784792, VIBA-5 y VIBA-6. Las alineaciones llevadas a cabo con alineaciones múltiples Clustal W Multiple Alignments comprendían todas estas nuevas cepas y empleaban 1434 nucleótidos de VIBA-1, 1279 de VIBA-2, 1252 de VIBA-3, 1432 de VIBA-4 y 1261 de VIBA-5 y 6. El análisis Blast N reveló que todos los aislamientos pertenecían a linajes rizobiales de α-Proteobacteria. La cepa cercana a VIBA-1 con una homología del 99,80 % fue Bradyrhizobium liaoningense LYG2, (número de acceso DQ497619), mientras que la especie tipo/cepa más cercana fue B. liaoningense USDA 3622 (acceso AF208513) con un 99,70 % de similitud. Los aislamientos VIBA-2 y VIBA-3 presentaron una similitud de 99,90 y 99,80, respectivamente, con Bradyrhizobium yuanmingense TSC10 (FJ540961), mientras que la homología con la especie tipo/cepa B. yuanmingense CCBAU 10071 (AF193818) fue 99,53 y 99,52 %, respectivamente. De la misma manera, VIBA-5 y 6 presentaron 99,30 % de similitud con Bradyrhizobium yuanmingense M11 (número de acceso AB601666) y una homología de 98,95 % con la especie tipo/cepa Bradyrhizobium yuanmingense CCBAU 10071, (número de acceso AF193818). Por otro lado, VIBA-4 tuvo una cercanía del 100 % con Agrobacterium sp. JS71 (número de acceso AY174112) y 99,10 % con el tipo de especie/cepa Rhizobium radiobacter (anteriormente Agrobacterium tumefaciens) NCPPB2437 (número de acceso D14500).
ANÁLISIS FILOGENÉTICO
El análisis filogenético indicó una relación evolutiva entre los aislamientos y la colección de cepas más importante (Figura 3).
La cepa VIBA-1 se localizó en la misma rama con la especie tipo/cepa Bradyrhizobium liaoningense, USDA 3622, mientras que VIBA-2, VIBA-3 y VIBA-5 y 6 se localizaron en la misma rama de Bradyrhizobium yuanmingense, CCBAU 10071, que es similar a los resultados del genotipo ARDRA.
DISCUSIÓN
El hecho de que las cepas nativas crecieron en medio de cultivo salino (6,6 dS m-1 de NaCl) implica algunas características de adaptación al estrés salino, aunque su crecimiento fue obviamente menor que la especie tipo/cepa CCBAU 10071. De hecho, la tolerancia a la sal de estas cepas fue menor que en otros informes, incluso se aisló de los mismos suelos cubanos notificados como altamente salinos (Babiney con EC 4,76 dS m-1) y muy altamente salinos (Jiguaní 5,8 dS m-1) (19). Con respecto a las especies de rizobios, se ha documentado el alto nivel de tolerancia a la sal de Phyllobacterium myrsinacearum, que puede crecer a un 2,5 a 3 % de salinidad (11). Mientras que Mesorhizobium ciceri (cepa GA-2) y Ensifer (Sinorhizobium) meliloti (cepas AFL-2 y AFL-3) crecieron a 1,5 y 2 % de los niveles de sal, al contrario que Bradyrhizobium liaoningense y Bradyrhizobium canariense, mostrando un verdadero desarrollo solo al 0,1 % del nivel de sal (11).
Sin embargo, Bradyrhizobium japonicum (E109) puede crecer en medios con concentraciones de NaCl de hasta 75 mM, lo que demuestra que la sensibilidad a la sal en esta especie se ve afectada por la fase de crecimiento del cultivo y el tipo de prueba utilizada para medir la tolerancia a la sal (20,21).
Otro tipo de estrés ampliamente conocido es la variación del pH (ácido o alcalino), que puede provocar daños en las bacterias rizobiales, principalmente en la supervivencia, la capacidad de infección y su diversidad (11,22). Nuestros resultados confirman estos hallazgos previos; debido a que las cepas no fueron capaces de desarrollarse en medio ácido (pH 4,5), pero al contrario, todos los aislados crecieron en condiciones alcalinas (pH 9).
Aunque estos resultados parecen ser la tendencia, las bacterias rizobiales adaptadas a ambientes ácidos, como Bradyrhizobium canariense, aislado de suelos ácidos de España (pH 5,3 y 4,8), han sido informadas por otros autores (11). Además, hay informes para la adaptación de rizobios a condiciones alcalinas también; incluso los aislados tienen la tendencia a ser más tolerantes a la alcalinidad que la acidez (22). Sin embargo, hay diferencias entre los géneros. Varios informes revelaron que Bradyrhizobium sp., es más tolerante a los ácidos que Rhizobium sp. Sin embargo, también se ha demostrado su capacidad para crecer en medio alcalino (23,24). A pesar de ello, se han encontrado y caracterizado cepas de Bradyrhizobium con tolerancia a pH 11 (25), mientras que otras, obtuvieron crecimiento en la mayoría de los aislamientos a pH 9 y unas pocas cepas alcanzaron su desarrollo a pH 4 (11).
Una posible explicación de la tolerancia a la acidez o alcalinidad se basa en la capacidad del microsimbionte para mantener el pH interno cerca de la neutralidad, que podría estar relacionado con la exclusión de protones, aumentar la capacidad del tampón citoplasmático y mantener altas concentraciones de potasio y glutamato. (26,27). Sin embargo, el crecimiento demostrado de estas cepas a pH alcalino es una buena respuesta ya que los suelos del valle del río Cauto son ligeramente alcalinos (9).
Por otro lado, los resultados de las variaciones de temperatura indican que las cepas no solo tienen la capacidad de desarrollarse en ambientes salinos y variaciones de pH, sino que también tienen capacidad para soportar altas temperaturas. La adaptación a alta temperatura de estas cepas puede estar relacionada con la región donde fueron aisladas, que tiene largos períodos de sequía y altas temperaturas (28,29), siendo esta una buena característica, con el fin de obtener una mejora del proceso simbiótico en plantas de caupí (30-32). Asimismo, otros autores han encontrado una alta variabilidad en la capacidad de crecimiento con temperaturas superiores a 35 oC, aunque la tendencia fue a disminuir en el número de células (30,33).
Los resultados de esta experiencia fueron similares a los obtenidos por otros investigadores, que aislaron y caracterizaron Bradyrhizobium sp. cepas de Centrosema sp., que crecieron a 40 oC (29).
En este estudio, logramos el aislamiento de cuatro cepas nativas de Bradyrhizobium a partir de nódulos de plantas de caupí cultivadas en suelo afectado por estrés salino. Sin embargo, solo dos especies diferentes fueron representadas por las cepas de Bradyrhizobium aisladas, que se obtuvieron a partir de la secuencia de 16S ADNr. El aislado VIBA-1 se localizó en especies de Bradyrhizobium liaoningense, mientras que VIBA-2, VIBA-3, VIBA-5 y VIBA-6 en especies de Bradyrhizobium yuanmingense. Sin embargo, VIBA-4 se identificó como R. radiobacter (anteriormente Agrobacterium tumefaciens).
El hecho de que solo se encontraron dos especies de Bradyrhizobium en nuestra investigación muestra una baja diversidad del género Bradyrhizobium en esta región. La reducción de la población bacteriana en suelos salinos puede atribuirse al escaso uso de especies leguminosas (29-33). La búsqueda y selección de nuevos aislamientos representados por un número mayor de especies rizobiales es necesaria para mejorar las poblaciones bacterianas y la calidad de la simbiosis.La especie Bradyrhizobium liaoningense se aisló inicialmente de las plantas de soja (34), mientras que Bradyrhizobium yuanmingense se aisló de los nódulos de Lespedeza (35). Esto mostró la capacidad de formar nódulos en Medicago sativa y Melilotus albus, mientras que no fue posible encontrar nódulos en la soja.
Sin embargo, se ha encontrado que B. yuanmingense es capaz de infectar raíces y establecer simbiosis en plantas de caupí, en contraste con B. liaoningense que no fue capaz de producir nódulos (36). En Cuba, estas especies no han sido reportadas en plantas de caupí cultivadas bajo condiciones de suelo salino. Sin embargo, B. liaoningense y B. yuanmingense se han aislado de suelos salinos, alcalinos y sódicos en seco (37,38).
También se aisló una cepa de Rhizobium radiobacter (anteriormente Agrobacterium tumefaciens, VIBA-4) de los nódulos muestreados; sin embargo, este aislado no fue capaz de formar estructuras nodulares en plantas de caupí cuando éstas fueron inoculadas nuevamente. Esta especie también ha sido aislada de nódulos de Retama sphaerocarpa (11); sin embargo, las cepas de A. tumefaciens pierden la capacidad de formar nódulos, lo que indica que este tipo de cepas puede pertenecer a una procesión de bacterias endofíticas que se deslizan en nódulos para protección (39, 40).
Varios hallazgos han reportado la presencia de otros microorganismos en los nódulos como proteobacterias no simbióticas, bacterias desnitrificantes, patógenas y fototrópicas de numerosos géneros entre los que se encuentra el Agrobacterium (11,41,42). Del mismo modo, aislados de las cepas de Agrobaterium se obtuvieron de nódulos de diferentes leguminosas, pero la mayoría de las cepas aisladas de Agrobacterium no pudieron nodular en sus hospederos originales, debido a que no se hibridaron con sondas génicas nif y nod al verificar que no eran bacterias simbióticas. (43-45). No obstante, otros autores han informado que algunas cepas de Agrobacterium podrían formar nódulos efectivos en algunas plantas leguminosas (46-48).
La especie Bradyrhizobium yuanmingense utilizada en los experimentos fue aislada en primer lugar de los nódulos de Lespedeza cuneata en el norte de China (36). Esta especie se ha aislado en una amplia gama de regiones de temperatura, regiones secas y cálidas del sudeste asiático y el sur de África, así como de diferentes plantas hospedadoras. Por esta razón, esta cepa se ha utilizado como referencia para identificar nuevos aislamientos, así como para investigar la adaptación de cepas en condiciones de estrés, debido a sus características tolerantes y su fácil adaptación a bajas y altas temperaturas, pH diferente y estrés hiperosmótico (49 -52).
Por último, los resultados demostraron que en los suelos afectados por la sal del valle del río Cauto, existen cepas nativas de Bradyrhizobium capaces de crecer bajo diferentes condiciones de estrés como la salinidad, el pH y la temperatura alta. Esto significa la posibilidad de utilizar nuevos inoculantes de cepas adaptadas a estas condiciones ambientales y mejorar la fijación simbiótica de nitrógeno en caupí (9).
Además, el hecho de que solo se encontraron dos especies de bradirizobios (B. yuanmingense y
B. liaoningense) pone énfasis en la necesidad de encontrar y caracterizar nuevas cepas de rizobios de los suelos salinos del Valle del río Cauto con el fin de aumentar la diversidad.
CONCLUSIONES
Se aislaron y caracterizaron seis cepas de rizobios de los géneros de Bradyrhizobium a partir de nódulos de frijol cultivado en suelos salinos del valle del río Cauto. Bradyrhizobium liaoningense y Bradyrhizobium yuanmingense fueron las especies representativas, que se informaron por primera vez en estas áreas.
Las cepas mostraron buenas respuestas a diferentes estreses abióticos (salinidad, alta temperatura y pH alcalino) que señalan su adaptación a las condiciones ambientales.
La creación de la primera colección de cepas de Bradyrhizobium adaptadas a los suelos del valle del río Cauto es un importante éxito para lograr nuevos inoculantes para mejorar la fijación del nitrógeno simbiótico y, en consecuencia, mejorar el rendimiento de las plantas de caupí.
Esta investigación es un paso más allá para obtener nuevos bioinoculantes/biofertilizantes para dinamizar los suelos salinos cubanos y contribuir al desarrollo de la agricultura orgánica en esta región.
AGRADECIMIENTOS
Este trabajo se llevó a cabo como parte del proyecto AECID, referencia A/8500/07 y A/019119/08, y desarrollado entre CSIC, (Madrid, España) y la Universidad de Granma, Cuba. BRD fue respaldado por los contratos de la Junta de Comunidades de Castilla-La Mancha (POII09-0182-3834 y POII10-0211-5015).
BIBLIOGRAFÍA
1. Bui EN. Soil salinity: A neglected factor in plant ecology and biogeography. Journal of Arid Environments. 2013;92 (Supplement C):14–25. doi:10.1016/j.jaridenv.2012.12.014
2. Setia R, Gottschalk P, Smith P, Marschner P, Baldock J, Setia D, et al. Soil salinity decreases global soil organic carbon stocks. Science of The Total Environment. 2013;465:267–72. doi:10.1016/j.scitotenv.2012.08.028
3. González LM, González MC, Ramírez R. Aspectos generales sobre la tolerancia a la salinidad en las plantas cultivadas. Cultivos tropicales. 2002;23(2):27–37.
4. Miao Y, Liao R, Zhang X-X, Liu B, Li Y, Wu B, et al. Metagenomic insights into salinity effect on diversity and abundance of denitrifying bacteria and genes in an expanded granular sludge bed reactor treating high-nitrate wastewater. Chemical Engineering Journal. 2015;277(Supplement C): 116–23. doi:10.1016/j.cej.2015.04.125
5. Sall SN, Ndour NYB, Diédhiou-Sall S, Dick R, Chotte J-L. Microbial response to salinity stress in a tropical sandy soil amended with native shrub residues or inorganic fertilizer. Journal of Environmental Management. 2015;161:30–7. doi:10.1016/j.jenvman.2015.06.017
6. Assefa T, Beebe SE, Rao IM, Cuasquer JB, Duque MC, Rivera M, et al. Pod harvest index as a selection criterion to improve drought resistance in white pea bean. Field Crops Research. 2013;148(Supplement C):24–33. doi:10.1016/j.fcr.2013.04.008
7. Gogile A, Andargie M, Muthuswamy M. Screening selected genotypes of cowpea [Vigna unguiculata (L.) Walp.] for salt tolerance during seedling growth stage. Pakistan Journal of Biological Sciences. 2013;16(14):671–679.
8. Soares BL, Ferreira PAA, Oliveira-Longatti SM de, Marra LM, Rufini M, Andrade MJB de, et al. Cowpea symbiotic efficiency, pH and aluminum tolerance in nitrogen-fixing bacteria. Scientia Agricola. 2014;71(3):171–80. doi:10.1590/S0103-90162014000300001
9. Gómez-Padilla E, Ruiz-Díez B, Fernández-Pascual M, López-Sánchez R, Bloem E, Eichler-Löbermann B. Inoculation with Native Bradyrhizobia Strains Improved Growth of Cowpea Plants Cultivated on a Saline Soil. Communications in Soil Science and Plant Analysis. 2016;47(19):2218–24. doi:10.1080/00103624.2016.1228950
10. Vincent JM. A Manual for the Practical Study of Root-nodule Bacteria [Internet]. Oxford: Blackwell Scientific Publications; 1970 [cited 2017 Sep 15]. 164 p. (Handbook). Available from: https://books.google.com.cu/books?id=dcQcAQAAIAAJ
11. Ruiz-Díez B, Fajardo S, Puertas-Mejía MA, Felipe M del R de, Fernández-Pascual M. Stress tolerance, genetic analysis and symbiotic properties of root-nodulating bacteria isolated from Mediterranean leguminous shrubs in Central Spain. Archives of Microbiology. 2009;191(1):35–46. doi:10.1007/s00203-008-0426-y
12. Weisburg WG, Barns SM, Pelletier DA, Lane DJ. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 1991;173(2):697–703. doi:10.1128/jb.173.2.697-703.1991
13. Lane DJ. 16S rRNA sequencing. In: Stackebrandt E, Goodfellow M, editors. Nucleic Acid Techniques in Bacterial Systematics (Modern Microbiological Methods) [Internet]. New York - Brisbane - Toronto - Singapore: John Wiley & Sons; 1991 [cited 2017 Sep 15]. p. 115–75. Available from: http://doi.wiley.com/10.1002/jobm.3620310616
14. Tamura K, Dudley J, Nei M, Kumar S. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) Software Version 4.0. Molecular Biology and Evolution. 2007;24(8):1596–9. doi:10.1093/molbev/msm092
15. Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution. 1987;4(4):406–25. doi:10.1093/oxfordjournals.molbev.a040454
16. Tukey JW. Comparing Individual Means in the Analysis of Variance. Biometrics. 1949;5(2):99–114. doi:10.2307/3001913
17. StatSoft. STATISTICA (data analysis software system) [Internet]. Version 10. US: StatSoft, Inc.; 2011. Available from: http://www.statsoft.com
18. Nasser AM, Hayat Q, Hayat S, Faizan M, Faraz A. Exogenous proline application enhances the efficiency of nitrogen fixation and assimilation in chickpea plants exposed to cadmium. Legume Research-An International Journal. 2016;39(2):221–7. doi:10.18805/lr.v0iOF.9291
19. López SRC, Samson R, Van Damme P, Eichler-Löbermann B, Gomez Padilla E. Response of Rhizobium - Clitoria ternatea combinations under salt stress in the Cauto Valley in Cuba. Revista Mexicana de Ciencias Pecuarias. 2011;2(2):199–207.
20. Deb K, Deb B, Pandey P. Isolation and characterization of root nodule bacteria associated with Cassia alata of Southern parts of Assam, India. International Journal of Pure & Applied Bioscience. 2015;3(1):58–63.
21. Guimarães AA, Florentino LA, Almeida KA, Lebbe L, Silva KB, Willems A, et al. High diversity of Bradyrhizobium strains isolated from several legume species and land uses in Brazilian tropical ecosystems. Systematic and Applied Microbiology. 2015;38(6):433–41. doi:10.1016/j.syapm.2015.06.006
22. Hungria M, Chueire LM de O, Coca RG, Megı́as M. Preliminary characterization of fast growing rhizobial strains isolated from soyabean nodules in Brazil. Soil Biology and Biochemistry. 2001;33(10):1349–61. doi:10.1016/S0038-0717(01)00040-2
23. Singh A. Soil salinization and waterlogging: A threat to environment and agricultural sustainability. Ecological Indicators. 2015;57(Supplement C):128–30. doi:10.1016/j.ecolind.2015.04.027
24. Wendt D, Duran R. Native bradyrhizobial symbionts of Lupinus mariae-josephae, a unique endemism thriving in alkaline soils in Eastern Spain [Internet] [phd]. [Valenciana, España]: E.T.S.I. Agrónomos (UPM); 2015 [cited 2017 Sep 15]. 192 p. Available from: http://oa.upm.es/37229/
25. Bécquer CJ, Prévost D, Cloutier J. Aspectos fisiológicos y genotípicos en rizobios aislados de leguminosas forrajeras. Pastos y Forrajes. 2001;24(2):123–131.
26. Lorite MJ, Muñoz S, Olivares J, Soto MJ, Sanjuán J. Characterization of Strains unlike Mesorhizobium loti That Nodulate Lotus spp. in Saline Soils of Granada, Spain. Applied and Environmental Microbiology. 2010;76(12):4019–26. doi:10.1128/AEM.02555-09
27. Hungria M, Menna P, Delamuta JRM. Bradyrhizobium, the ancestor of all rhizobia: phylogeny of housekeeping and nitrogen-fixation genes. Biological Nitrogen Fixation. 2015;2:191–202. doi:10.1002/9781119053095.ch18
28. Grönemeyer JL, Kulkarni A, Berkelmann D, Hurek T, Reinhold-Hurek B. Rhizobia Indigenous to the Okavango Region in Sub-Saharan Africa: Diversity, Adaptations, and Host Specificity. Applied and Environmental Microbiology. 2014;80(23):7244–57. doi:10.1128/AEM.02417-14
29. Suzuki Y, Adhikari D, Itoh K, Suyama K. Effects of temperature on competition and relative dominance of Bradyrhizobium japonicum and Bradyrhizobium elkanii in the process of soybean nodulation. Plant and Soil. 2014;374(1–2):915–24. doi:10.1007/s11104-013-1924-5
30. Mathu S, Herrmann L, Pypers P, Matiru V, Mwirichia R, Lesueur DD. Potential of indigenous bradyrhizobia versus commercial inoculants to improve cowpea (Vigna unguiculata L. walp.) and green gram (Vigna radiata L. wilczek.) yields in Kenya. Soil Science and Plant Nutrition. 2012;58(6):750–63. doi:10.1080/00380768.2012.741041
31. Win KT, Oo AZ. Genotypic difference in salinity tolerance during early vegetative growth of cowpea (Vigna unguiculata L. Walp.) from Myanmar. Biocatalysis and Agricultural Biotechnology. 2015;4(4):449–55. doi:10.1016/j.bcab.2015.08.009
32. Nehra V, Choudhary M. A review on plant growth promoting rhizobacteria acting as bioinoculants and their biological approach towards the production of sustainable agriculture. Journal of Applied and Natural Science. 2015;7(1):540–556.
33. Lira MA, Nascimento LRS, Fracetto GGM. Legume-rhizobia signal exchange: promiscuity and environmental effects. Frontiers in Microbiology. 2015;6(945):1–9. doi:10.3389/fmicb.2015.00945
34. Xu LM, Ge C, Cui Z, Li J, Fan H. Bradyrhizobium liaoningense sp. nov., Isolated from the Root Nodules of Soybeans. International Journal of Systematic and Evolutionary Microbiology. 1995;45(4):706–11. doi:10.1099/00207713-45-4-706
35. Yao ZY, Kan FL, Wang ET, Wei GH, Chen WX. Characterization of rhizobia that nodulate legume species of the genus Lespedeza and description of Bradyrhizobium yuanmingense sp. nov. International Journal of Systematic and Evolutionary Microbiology. 2002;52(6):2219–30. doi:10.1099/00207713-52-6-2219
36. Steenkamp ET, Stępkowski T, Przymusiak A, Botha WJ, Law IJ. Cowpea and peanut in southern Africa are nodulated by diverse Bradyrhizobium strains harboring nodulation genes that belong to the large pantropical clade common in Africa. Molecular Phylogenetics and Evolution. 2008;48(3):1131–44. doi:10.1016/j.ympev.2008.04.032
37. Bhattacharya C, Pandey B. Isolation & Characterization of Rhizobium Species and its Effect on Growth on Monocot Plant used as Biofertilizer. International Journal of Research. 2015;2(1):597–604.
38. Land PE, Shutler JD, Findlay HS, Girard-Ardhuin F, Sabia R, Reul N, et al. Salinity from Space Unlocks Satellite-Based Assessment of Ocean Acidification. Environmental Science & Technology. 2015;49(4):1987–94. doi:10.1021/es504849s
39. Benson O, Beatrice A, Regina N, Koech PK, Skilton RA, Francesca S. Morphological, genetic and symbiotic characterization of root nodule bacteria isolated from Bambara groundnuts (Vigna subterranea L. Verdc) from soils of Lake Victoria basin, western Kenya. Journal of Applied Biology and Biotechnology. 2015;3(1):1–10.
40. Chihaoui S-A, Trabelsi D, Jdey A, Mhadhbi H, Mhamdi R. Inoculation of Phaseolus vulgaris with the nodule-endophyte Agrobacterium sp. 10C2 affects richness and structure of rhizosphere bacterial communities and enhances nodulation and growth. Archives of Microbiology. 2015;197(6):805–13. doi:10.1007/s00203-015-1118-z
41. Youseif SH, Abd El-Megeed FH, Ageez A, Mohamed ZK, Shamseldin A, Saleh SA. Phenotypic characteristics and genetic diversity of rhizobia nodulating soybean in Egyptian soils. European Journal of Soil Biology. 2014;60(Supplement C):34–43. doi:10.1016/j.ejsobi.2013.10.008
42. Zgadzaj R, James EK, Kelly S, Kawaharada Y, de Jonge N, Jensen DB, et al. A Legume Genetic Framework Controls Infection of Nodules by Symbiotic and Endophytic Bacteria. McDowell JM, editor. PLOS Genetics. 2015;11(6):e1005280. doi:10.1371/journal.pgen.1005280
43. Tariq M, Hameed S, Yasmeen T, Zahid M, Zafar M. Molecular characterization and identification of plant growth promoting endophytic bacteria isolated from the root nodules of pea (Pisum sativum L.). World Journal of Microbiology and Biotechnology. 2014;30(2):719–25. doi:10.1007/s11274-013-1488-9
44. Xu L, Zhang Y, Wang L, Chen W, Wei G. Diversity of endophytic bacteria associated with nodules of two indigenous legumes at different altitudes of the Qilian Mountains in China. Systematic and Applied Microbiology. 2014;37(6):457–65. doi:10.1016/j.syapm.2014.05.009
45. Ormeño-Orrillo E, Servín-Garcidueñas LE, Rogel MA, González V, Peralta H, Mora J, et al. Taxonomy of rhizobia and agrobacteria from the Rhizobiaceae family in light of genomics. Systematic and Applied Microbiology. 2015;38(4):287–91. doi:10.1016/j.syapm.2014.12.002
46. Alías-Villegas C, Cubo MT, Lara-Dampier V, Bellogín RA, Camacho M, Temprano F, et al. Rhizobial strains isolated from nodules of Medicago marina in southwest Spain are abiotic-stress tolerant and symbiotically diverse. Systematic and Applied Microbiology. 2015;38(7):506–14. doi:10.1016/j.syapm.2015.07.003
47. Karmakar K, Rana A, Rajwar A, Sahgal M, Johri BN. Legume-Rhizobia Symbiosis Under Stress. In: Plant Microbes Symbiosis: Applied Facets [Internet]. New Delhi: Springer; 2015 [cited 2017 Sep 15]. p. 241–58. doi:10.1007/978-81-322-2068-8_12
48. Oliveira-Longatti SM de, Marra LM, Soares BL, Bomfeti CA, Silva K da, Ferreira PAA, et al. Bacteria isolated from soils of the western Amazon and from rehabilitated bauxite-mining areas have potential as plant growth promoters. World Journal of Microbiology and Biotechnology. 2014;30(4):1239–50. doi:10.1007/s11274-013-1547-2
49. Narula S, Anand RC, Dudeja SS, Kumar V, Pathak DV. Molecular diversity of root and nodule endophytic bacteria from field pea (Pisum sativum L.). Legume Research: An International Journal. 2013;36(4):344–50.
50. Giri R, Dudeja SS. Host specificity of plant endophytic bacterial interactions: Root and nodule colonization under sterilized sand conditions in disposable coffee cups. Central European Journal of Experimental Biology. 2013;2(4):22–26.
51. Jha PN, Gupta G, Jha P, Mehrotra R. Association of rhizospheric/endophytic bacteria with plants: a potential gateway to sustainable agriculture. Greener Journal of Agricultural Sciences. 2013;3(2):73–84.
52. Kumar V, Pathak DV, Dudeja SS, Saini R, Giri R, Narula S, et al. Legume nodule endophytes more diverse than endophytes from roots of legumes or non legumes in soils of Haryana, India. Journal of Microbiology and Biotechnology Research. 2013;3(3):83–92.
Recibido: 27/10/2016
Aceptado: 24/04/2017
Ernesto Gómez Padilla, Centro de Estudio de biotecnología Vegetal, Facultad de Ciencias Agrícolas, Universidad de Granma, Cuba.Email: egomezp@udg.co.cu