INTRODUCCIÓN
La Fijación Biológica del Nitrógeno (FBN) es un proceso biológico de suma importancia pues constituye la única vía, hasta ahora conocida, donde la mayor reserva de nitrógeno que existe en el planeta (N2 (g)) se reduce a amonio, un estado químico que puede ser asimilado por las plantas 1. Este proceso solo se realiza por un grupo de procariotas (bacterias y archeas), denominadas diazótrofas 2.
Los rizobios son las Bacterias Promotoras del Crecimiento Vegetal (BPVC) que mayor contribución realizan a la FBN en la zona terrestre del planeta. Los estudios sobre la asociación simbiótica de estas bacterias con las plantas leguminosas datan de al menos 125 años y han comprobado que dicha interacción aporta alrededor de 600 kg N ha-1 año-1 (3,4. Sin embargo, la rizosfera y endosfera de algunas gramíneas de importancia económica como el maíz (Zea mays L.) y el arroz (Oryza sativa L.), también se han descrito como hábitats de estas bacterias, por lo que desde hace algunos años se busca extender a las gramíneas los conocimientos y usos de la FBN que se conoce en las leguminosas 5-7.
Por otra parte, existen diferentes métodos moleculares, fisiológicos y ecológicos que permiten clasificar a una BPCV como diazotrófica. Entre ellos se encuentran la amplificación del gen nifH 8, el crecimiento de las cepas bacterianas en medios semisólidos libre de nitrógeno 9, el ensayo de reducción del acetileno (ARA, por sus siglas en inglés) 10, los ensayos de inoculación de bacterias en plantas leguminosas patrones 11 y las técnicas isotópicas con N15 (12. Específicamente en plantas leguminosas inoculadas con estas bacterias fijadoras de nitrógeno se puede determinar, además, la actividad de la enzima glutamina sintetasa en nódulos y la concentración de ureidos en nódulos y hojas de las plantas 13.
Varios estudios, en los que se han empleado algunos de los métodos mencionados anteriormente, defienden la hipótesis de que la fitoestimulación es el principal mecanismo que emplean los rizobios para promover el crecimiento del arroz y no la FBN 14. Son escasas las evidencias que muestran la presencia de rizobios fijadores de nitrógeno en ecosistemas donde no hayan estado presentes leguminosas como cultivos precedentes a las gramíneas. En Cuba se carece de estudios donde se determine la capacidad para fijar nitrógeno de cepas de rizobios que se asocien, de manera natural, a gramíneas de importancia económica como el arroz. Por lo tanto, el objetivo de esta investigación fue evaluar la capacidad para fijar nitrógeno de cepas de rizobios, provenientes de la rizosfera de plantas de arroz cultivares INCA LP-5 e INCA LP-7, cultivados en monocultivo.
MATERIALES Y MÉTODOS
La investigación se realizó en el año 2018, en el Instituto Nacional de Ciencias Agrícolas (INCA) de Cuba y en el Centro de Ciencias Genómicas de la Universidad Nacional Autónoma de México.
Material biológico
Oligonucleótidos
Se emplearon tres pares de oligonucleótidos degenerados para la amplificación del gen nifH (Tabla 1). Estos cebadores se diseñaron teniendo en cuenta secuencias del gen nifH de 150 proteobacterias, provenientes de nódulos de diferentes leguminosas 15.
Tabla 1.
Oligonucleótidos que se emplearon en la amplificación del gen nifH de las tres cepas de rizobios
Cepas bacterianas
Se emplearon tres cepas de rizobios pertenecientes al cepario del Laboratorio de Microbiología del Departamento de Fisiología y Bioquímica Vegetal del INCA. Dos cepas: Rhizobium sp. Rpr11 (Rpr11) y Rhizobium sp. Rpd16 (Rpd16), provinieron de la rizosfera de plantas de arroz cultivar INCA LP-5 y la cepa Rhizobium sp. 5P1 (5P1) de la rizosfera del cultivar INCA LP-7 16. En el momento del aislamiento de estas cepas, ambos cultivares se encontraban establecidos en un suelo Gleysol Nodular Ferruginoso Petroférrico del municipio “Los Palacios”, provincia Pinar del Río, Cuba. Este suelo se caracteriza por su baja fertilidad natural, debido a su explotación con monocultivo intensivo de arroz durante 50 años 17. Además, se utilizaron dos cepas como controles positivos de referencia: Rhizobium etli CFN42 (CFN42) para detectar el gen nifH 18 y la cepa Bradyrhizobium elkanii ICA 8001 (ICA 8001) en un ensayo de crecimiento en medios libre de nitrógeno 19.
Se obtuvieron inóculos de las cinco cepas de rizobios, a partir de la inoculación de una asada de éstas en frascos Erlenmeyers de 100 mL, que contenían 10 mL del medio Triptona-Extracto de levadura (TY) 20. Los Erlenmeyers se mantuvieron en agitación a 150 r min-1 y 30 ºC, durante 16 horas. La pureza de los inóculos se monitoreó mediante tinción de Gram. La concentración celular se determinó mediante el método de las diluciones seriadas y el cultivo en placas Petri con medio TY sólido; las cuales se incubaron durante 48 h a 30 ºC. La concentración de rizobios en los inóculos se ajustó a 5 x 109 UFC mL-1.
Semillas de leguminosas, desinfección y germinación
Se emplearon semillas de siratro (Macroptillium atropurpureum) y soya (Glycine max L.) en ensayos de inoculación en condiciones controladas. Las semillas se desinfectaron superficialmente y se pre germinaron. Las de siratro se mantuvieron en etanol al 70 % durante 5 min, posteriormente se lavaron con agua destilada y se sometieron a un tratamiento con ácido sulfúrico concentrado durante 10 min. Se sumergieron en hipoclorito de sodio al 25 % (v/v) durante 15 min y se lavaron 10 veces con agua destilada estéril. Para la desinfección de las semillas de soya, se sumergieron en etanol al 70 % durante 5 min y posteriormente en hipoclorito de sodio al 25 % (v/v) por 15 min. Por último, se lavaron 10 veces con agua destilada estéril. Ambos grupos de semillas se colocaron en placas con agar agua (0,75 %) (m/v) y se incubaron a 30 °C en la oscuridad para favorecer la germinación. Las semillas de siratro se mantuvieron por 24 h en esta condición mientras que, las de soya estuvieron durante 72 h.
Detección de genes nifH en las cepas de Rhizobium
A partir de inóculos de las cepas de rizobios Rpr11, Rpd16, 5P1 y CFN42, se realizó la extracción del ADN genómico bacteriano mediante el UltraClean® Microbial DNA Isolation Kit, de MOBIO Laboratories, Inc. Para comprobar el éxito de la extracción, 5 µL del extracto se analizó mediante electroforesis en gel de agarosa 1,5 % (m/v) en TAE 1X. El tiempo y voltaje de la corrida electroforética, así como el marcador de peso molecular que se emplearon, se describieron previamente 15.
La amplificación del gen nifH se realizó mediante PCR a partir de los extractos del ADN genómico. Como cebadores se emplearon tres pares de oligonucleótidos degenerados (Tabla 1), a una concentración de 0,01 mM y un volumen de 3 µL por reacción de PCR. Los restantes componentes de la mezcla de la PCR fueron: tampón de reacción Taq 10X, 5 µL; MgCl 25 mM, 7 µL; Dimetil sulfóxido (DMSO) 99,5 %, 1 µL; dNTPs 10 mM, 1 µL; Polimerasa Taq 1 U µL-1, 1 µL; DNA bacteriano 30 ng µL-1, 1,0 µL y agua ultra pura 34 µL; en un volumen final de 50 μL por reacción de PCR.
Las condiciones que se utilizaron para realizar la PCR fueron: 1 ciclo a 94 oC, 3 min; 30 ciclos con un gradiente de temperaturas de alineamiento de 62 oC a 56 oC, 15 seg; 72 oC de extensión por 1 min y un ciclo a 72 oC, 10 min. El producto del PCR se analizó mediante electroforesis en gel de agarosa 1,5 % (m/v) en TAE 1X. El tiempo y voltaje de la corrida electroforética, así como el marcador de peso molecular que se emplearon, se describieron en investigaciones previas 15.
Crecimiento de las cepas de Rhizobium en medios semisólidos libre de nitrógeno
A partir de los inóculos de las cepas Rpr11, Rpd16 y 5P1 se prepararon suspensiones celulares. Para ello, 1 mL de los inoculantes se centrifugó a 10 000 rpm, 10 min y se desechó el sobrenadante. El pellet se resuspendió en 1 mL de solución salina (NaCl, 0,9 % m/v) estéril. Este procedimiento se repitió dos veces para lograr una suspensión celular carente de los componentes del medio TY.
Se inocularon 100 μL de las suspensiones bacterianas en frascos que contenían 10 mL de los medios semisólidos libre de nitrógeno Rennie y JMV 21. La inoculación se realizó liberando la suspensión celular de la parte inferior del medio a la superficie con una micropipeta. El control negativo del experimento consistió en frascos donde se inocularon 100 μL de la solución salina estéril. El control positivo consistió en frascos que se inocularon con el mismo volumen y concentración de una suspensión celular de la cepa ICA 8001.
Los frascos se incubaron durante cinco días a 30 oC y se determinó la capacidad de crecimiento de las cepas bacterianas mediante la formación de halos opacos en la superficie y el interior del medio. La tendencia al amarillamiento del medio JMV se interpretó como la producción de ácidos orgánicos 21. El ensayo se realizó dos veces y se emplearon cinco frascos por cada una de las cepas bacterianas.
Nodulación de plantas leguminosas inoculadas con las cepas de Rhizobium
Para determinar la capacidad de formar nódulos efectivos en la FBN de las cepas Rpr11, Rpd16 y 5P1; se realizaron ensayos de inoculación en plantas de siratro y soya. Semillas germinadas de siratro, con raíces de aproximadamente 1-2 cm de largo se colocaron en frascos de 200 mL que contenían 50 mL de medio Norris y Date semisólido 22, a razón de una semilla por frasco. De manera similar se procedió con las semillas germinadas de soya, las cuales se colocaron en tubos de ensayo (diámetro 2 cm, largo 20 cm) que contenían 20 mL del mismo medio.
Posteriormente, se aplicaron sobre cada semilla de siratro y soya inóculos de los tres aislados de rizobios, a razón de 0,5 mL y 1,0 mL, respectivamente. Semillas de ambas leguminosas, que se inocularon con medio LM estéril, se emplearon como control negativo del ensayo. La investigación se realizó dos veces con un diseño Completamente Aleatorizado y se utilizaron siete plantas por tratamiento en siratro y nueve para soya.
Las plantas crecieron en condiciones controladas con un fotoperíodo de 12 h luz/12 h oscuridad, a una temperatura día/noche de 26/22 °C y humedad relativa del 70 %. Cinco semanas después de la inoculación, se determinó en ambas leguminosas: el número de nódulos en la raíz primaria; el número total de nódulos; la efectividad de los nódulos totales en la FBN y la masa seca de los nódulos totales. La efectividad de los nódulos se determinó mediante el método visual. Para ello, se realizó su disección con hojas de bisturí de acero inoxidable, detectando o no la presencia de una coloración rojiza en el interior, característico de la proteína leghemoglobina 23.
Análisis estadístico
Los datos provenientes de los ensayos de inoculación de las plantas de siratro y soya se sometieron a la prueba de normalidad (prueba de Bartlett) y homogeneidad de varianza (prueba de Kormogorov-Smirnov). Los datos de número de nódulos en la raíz principal, totales y efectivos totales, se transformaron por la raíz cuadrada del valor, previo a su procesamiento estadístico. Se aplicó análisis de varianza de clasificación simple, con la prueba de comparación de medias de Tukey con p<0,05; para determinar las diferencias entre las medias. Se utilizó el programa Statgraphic Plus versión 5.0 para el procesamiento estadístico de los datos.
RESULTADOS Y DISCUSIÓN
La mayoría de los trabajos que abordan la identificación de rizobios asociados a gramíneas, se han realizado en áreas experimentales sometidas a técnicas de rotación de cultivo con plantas leguminosas 24,25. Según estas investigaciones, las leguminosas incrementan las poblaciones de rizobios fijadores de nitrógeno y la gramínea se beneficia del nitrógeno remanente en el suelo proveniente de la FBN. En este sentido, se ha comprobado que la contribución en nitrógeno que hacían algunas cepas de rizobios al cultivo del arroz, no provenía de la FBN; sino del presente en el suelo donde se encontraba el trébol (Trifolium alexandrinum) como cultivo antecesor 14.
En la presente investigación se emplearon métodos moleculares y fisiológicos para determinar la capacidad de realizar la fijación de nitrógeno de tres cepas del género Rhizobium provenientes de la rizosfera de plantas de arroz cultivado en monocultivo intensivo. Se extrajo el ADN genómico de las cepas de rizobios en estudio, el cual se visualizó como bandas en la parte superior del gel de agarosa (Figura 1). Sin embargo, no se amplificó el gen nifH en las cepas de Rhizobium 5P1, Rpr11 y Rpd16, con ninguno de los tres pares de cebadores que se emplearon. En todos los geles se observó una banda de 300-400 pb correspondiente a la amplificación del gen nifH de la cepa de referencia CFN42 (Figura 2).
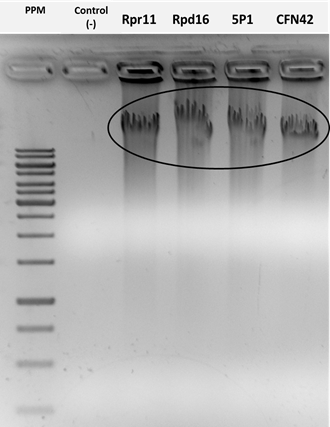
PPM, Patrón de Peso Molecular; Control (-), control negativo
Figura 1.
Electroforesis en gel de agarosa (1,5 %) (m/V) de los extractos de ADN de la cepa Rhizobium etli CFN42 y de tres aislados de rizobios, provenientes de los cultivares de arroz INCA LP-5 e INCA LP-7
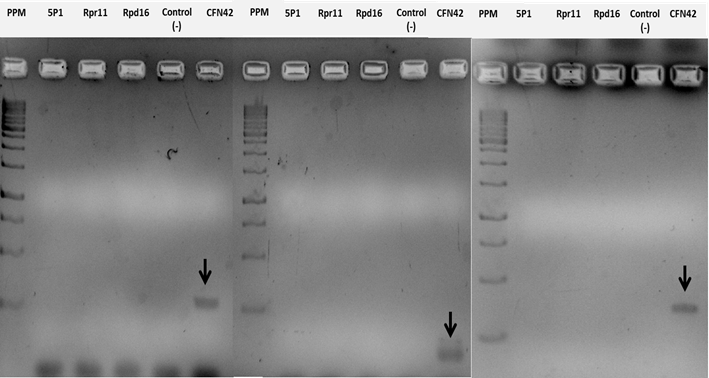
Los cebadores que se emplearon fueron nifH_AIF20187 y nifH_AIR2088 (A), nifH_UN_FW7807 y nifH_UN_RV7808 (B); KAD18 y GEM181 (C). PMM, Patrón de Peso Molecular; Control (-), Control negativo
Figura 2.
Electroforesis en gel de agarosa (1,5 %) (m/V) de los productos de PCR del gen nifH de la cepa de referencia Rhizobium etli CFN42 y de tres aislados de rizobios provenientes de los cultivares de arroz INCA LP-5 e INCA LP-7
Resultados similares, en cuanto a la amplificación del gen nifH, se obtuvieron en investigaciones anteriores con cepas de Rhizobium provenientes de la rizosfera de plantas de arroz 26. De los genes que codifican para el complejo de la nitrogenasa, la amplificación del gen nifH no solo se realiza para detectar la presencia de esta enzima en las bacterias, sino también para realizar estudios de filogenia con microorganismos fijadores de nitrógeno 2.
Se conoce que genes como el nifH necesitan amplificarse con oligonucleótidos que contengan altos niveles de degeneración y con temperaturas adecuadas en la PCR, que permitan la hibridación entre los cebadores y la región correspondiente del ADN bacteriano 8. Por ello, se emplearon oligonucleótidos degenerados y un gradiente considerable de temperaturas de alineamiento. Sin embargo, no se logró su amplificación en las cepas de Rhizobium.
Otro aspecto a considerar en este tipo de estudios es la secuencia de los juegos de cebadores. Para el caso de los genes nifH, se han desarrollado cebadores de PCR universales y específicos de grupo, con el propósito de amplificar diversas secuencias de este gen. Sin embargo, la mayoría de ellos solo amplifican el 35 % de secuencias dentro de las alfa, beta y gamma proteobacterias. Además, en el caso de los cebadores universales para el gen nifH, menos del 50 % han permitido detectar secuencias conocidas de este gen, debido a la especialmente notable variación en la cobertura de este tipo de cebadores. Incluso, estudios previos demostraron que en el caso particular de los cebadores universales PolF/PolR solo cubren el 25 % de la diversidad nifH presentes en bases de datos 8.
Por otra parte, la falta de especificidad de algunos de los cebadores de genes nifH han amplificado secuencias del ADN de cepas de Escherichia coli, bacterias que hasta ahora no se han descrito como diazotróficas. La dimerización del cebador, la formación de horquillas, el contenido de guanina y citosina y la termodinámica del cebador; constituyen factores que afectan la especificidad y la cobertura de los cebadores 8. Teniendo en cuenta todo lo anterior, pudiera pensarse no solo en aspectos de la técnica de amplificación de PCR que explicarían la no detección del gen nifH en los tres aislados de Rhizobium que se estudiaron, sino también la falta de especificidad y cobertura de los juegos de cebadores que se emplearon, independientemente de que fueran diseñados a partir de secuencias de genes nifH de bacterias diazotróficas. La no detección del gen nifH en el genoma bacteriano por el método empleado, no necesariamente significa que las cepas carezcan de la capacidad para realizar la FBN. Sería necesario emplear otros juegos de cebadores que podrían diseñarse a partir de secuencias provenientes de bacterias diazotróficas asociativas de gramíneas.
En esta investigación se evaluó la capacidad de tres cepas de Rhizobium para realizar la FBN, desde el punto de vista fisiológico. Se evidenció el crecimiento de estas bacterias en dos medios de cultivo libre de nitrógeno (JMV y Rennie), a los cinco días de incubación. En ningún caso se observó cambio en la coloración del medio JMV. En investigaciones previas se constató que cepas de Bacillus, Paenibacillus y Azospirillum; géneros que presentan una elevada frecuencia de aparición en la rizosfera de plantas de maíz y arroz; también tienen la capacidad de crecer en estos medios de cultivo 27,28.
Estudios recientes han demostrado la capacidad de cepas de rizobios provenientes de gramíneas para crecer en medios libre de nitrógeno. Tal es el caso de cepas de Bradyrhizobium asociadas a la rizosfera de sorgo (Sorghum bicolor (L.) Moench), las cuales crecieron en medio Rennie y cepas de Rhizobium undicola, endófitos de plantas de arroz con la capacidad de crecer en el medio JNFb 29,30. En ambos casos, se confirma la diazotrofía de las cepas mediante la técnica del ARA. Los medios libres de nitrógeno, como los que se emplearon en esta investigación, se utilizan en múltiples trabajos con la finalidad de seleccionar microorganismos diazotróficos y así descartar gran cantidad de microorganismos cultivables que se obtienen durante el proceso de aislamiento 9.
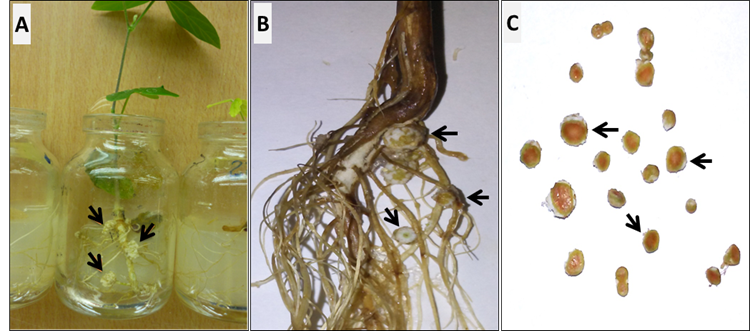
En esta investigación; además, se realizaron ensayos de inoculación en plantas de siratro y soya para evaluar la capacidad de las cepas de Rhizobium para realizar la FBN. Los resultados mostraron la formación de nódulos efectivos en ambas leguminosas, tras el empleo de las cepas bacterianas provenientes de la rizosfera de plantas de arroz (Figura 3, Tabla 2).
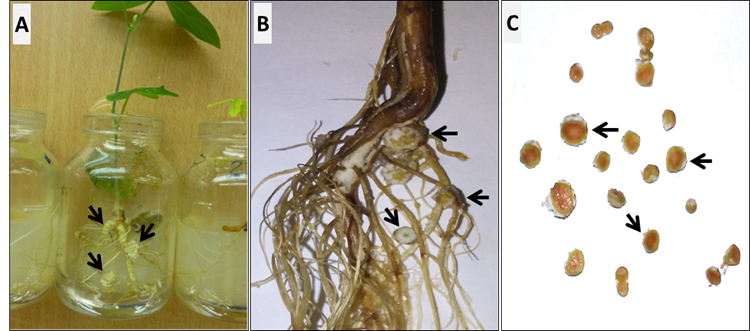
La coloración rojiza en el interior de los nódulos de soya (C) muestra su efectividad en la FBN. Los resultados corresponden a la primera repetición del experimento debido a su semejanza con los obtenidos en la segunda repetición
Figura 3.
Presencia de nódulos en las raíces de plantas de siratro (A) y soya (B) inoculadas con uno de los aislados de rizobios
Tabla 2.
Efecto de la inoculación de las cepas Rpd16, Rpr11 y 5P1 en la nodulación de plantas de siratro y soya
Los resultados corresponden a la primera repetición del experimento debido a su semejanza con los obtenidos en la segunda repetición.
NNrp, número de nódulos en la raíz primaria; NNt, número de nódulos totales; NNte, número de nódulos totales efectivos; MSNt, masa seca de nódulos totales. Control, plantas inoculadas con medio LM estéril. Media con letras iguales en la misma columna no difieren significativamente (Tukey α=0,05 y n=7(siratro), n=9 (soya))
Los resultados mostraron que la inoculación de la cepa Rpr11 en las plantas de siratro produjo los mayores valores en todas las variables de nodulación. En las plantas que se trataron con la cepa Rpd16 se observaron nódulos. Sin embargo, no existieron diferencias significativas con el tratamiento control en ninguna de las variables que se midieron. Siratro, constituye una leguminosa forrajera que se ha establecido como planta modelo para evaluar la capacidad de infección y la efectividad de la nodulación de aislados de rizobios en condiciones controladas, pues responde como hospedero a un amplio espectro de cepas de rizobios provenientes de diversos hábitats 31.
Independientemente de la versatilidad de leguminosas como siratro para nodular con diferentes géneros del grupo de los rizobios, se ha discutido sobre la especificidad de la planta y la bacteria para establecer la simbiosis 32,33. En este sentido, la soya; otra de las leguminosas empleadas en esta investigación, se asocia generalmente con especies del género Bradyrhizobium34. Sin embargo, investigaciones recientes demuestran que otros géneros como Rhizobium, Ensifer, Mesorhizobim y Sinorhizobium también forman nódulos en las raíces de estas plantas 35. La inoculación de la cepa 5P1 incrementó significativamente el número de nódulos de la raíz primaria de las plantas de soya; mientras que, el empleo de la cepa Rpr11 produjo los mayores valores en el número de nódulos totales, totales efectivos y la masa seca de los nódulos en estas plantas.
La disección de los nódulos presentes en las plantas de siratro y soya, que se inocularon con las tres cepas de Rhizobium, permitió observar una coloración rojiza en el interior de los mismos (Figura 3C). El color marrón, rojo o rosado es característico de la leghemoglobina, proteína indicadora de la efectividad de los nódulos en el proceso de FBN 23. Esta proteína tiene un grupo prostético tipo protohemo que se enlaza de forma reversible con el oxígeno una vez que este difunde hasta la zona central de los nódulos y lo transporta desde la membrana plasmática de las células infectadas hacia la membrana del simbiosoma. De esta manera, se establecen las concentraciones adecuadas de oxígeno en los nódulos para mantener viable al rizobio, proteger a la nitrogenasa de las altas concentraciones de oxígeno y favorecer la FBN 36.
Los resultados de esta investigación constituyen evidencias aparentemente contradictorias. Por un lado, el método molecular que se empleó no permitió amplificar el gen nifH. Sin embargo, los métodos fisiológicos como el crecimiento en medios semisólidos libre de nitrógeno y los ensayos de nodulación en plantas leguminosas, muestran que estas bacterias sí tienen la capacidad de fijar el nitrógeno del aire.
El empleo de métodos moleculares más sofisticados en los estudios taxonómicos de bacterias ha permitido reformular conceptos tradicionales para intentar caracterizar, clasificar y nombrar a cepas de rizobios. Estas bacterias Gram negativas se diferencian del resto de los procariotas por su capacidad de fijar nitrógeno y formar nódulos en las leguminosas 4. Sin embargo, estudios recientes donde se emplean secuenciación de ADN total, indican que ambos atributos no necesariamente son propiedades inherentes a los rizobios 37. Estas investigaciones concluyen que cepas del género Bradyrhizobium tienen una gran diversidad en cuanto a la presencia de los genes nif y nod, estos últimos con la información genética para la formación de los nódulos. Ese estudio contempla que algunas de las cepas de Bradyrhizobium presentan ambos grupos de genes, otras ninguno de ellos y otras cepas solo los nif; independientemente del sitio de donde fueron aisladas (suelo, nódulos de leguminosas). Por lo tanto, en el estudio de cepas de rizobios, enfocados a la capacidad de formar nódulos y fijar nitrógeno, tendría que tenerse en cuenta las particularidades genéticas descritas previamente.
Por otra parte, la capacidad de fijar nitrógeno en los rizobios tiene que contemplar la posibilidad de hacerlo, no solo en un contexto simbiótico con leguminosas, sino también en las otras formas de vida, en las que se han podido constatar que estos microorganismos pueden sobrevivir, dígase en vida libre, asociativa y endofítica 37. Las tres cepas que se estudiaron en esta investigación constituyen ejemplos de rizobios que, aislados de la rizosfera de dos cultivares de arroz de importancia económica (INCA LP-5 e INCA LP-7), cuentan con la capacidad de formar nódulos efectivos en plantas de siratro y soya. Sería necesario corroborar la capacidad de las cepas para fijar nitrógeno por métodos con mayor sensibilidad y especificidad como el ARA y el empleo del N15.
El hecho de encontrar cepas de rizobios en áreas sometidas a prácticas de monocultivo intensivo de arroz, que se asocian naturalmente a esta gramínea, en suelos caracterizados por su baja fertilidad y con potencialidades de fijar nitrógeno, brinda un importante valor práctico a esas cepas bacterianas. El empleo de formulados a base de estas bacterias pudiera formar parte del manejo integrado del cultivo, con un ahorro de fertilizantes químicos. Esta tecnología adquiere especial relevancia en el contexto de una agricultura ecológica y económicamente sostenible, sobre todo en cultivos de alta demanda y de grandes áreas cultivadas como el arroz, donde cada año se incrementan los rendimientos a costa del uso irracional de fertilizantes químicos, productos que afectan negativamente a los ecosistemas 5.
CONCLUSIONES
Cepas de Rhizobium, asociadas a plantas de arroz, cultivares INCA LP-5 e INCA LP-7 cultivadas en monocultivo, parecen tener la capacidad de fijar el nitrógeno del aire, a pesar de no mostrar amplificación del gen nifH. Si bien la presencia de este gen es una condición estrictamente necesaria para que una bacteria fije nitrógeno, también son necesarias ciertas condiciones ambientales que propicien este proceso. Por lo tanto, podrían modificarse técnicas ya establecidas e incluso implementar nuevas, que permitan evaluar con precisión la capacidad de fijar nitrógeno de los rizobios asociados a plantas no leguminosas.
Este estudio constituye un primer acercamiento a la capacidad de fijar nitrógeno de cepas de rizobios rizosféricos de arroz, cultivado en suelos expuestos al monocultivo intensivo de la gramínea.